MRI研究の裾野を広げていくことを目的に脳MRI解析チュートリアルを開催します。チュートリアルでは十分な解析手法を持たない国内各地の研究者を対象に、実際に脳MRI解析を体験してもらいながら実践的な脳MRI解析手法を実演、説明を行います。
2024年2月ABiS脳画像解析チュートリアル
2024年2月17日(土)~18日(日)、完全オンラインで「2024年2月ABiS脳画像解析チュートリアル」を開催致しました。
チュートリアルは盛況のうちに終了致しました。多数のご参加ありがとうございました。
研究職・医療職、大学教員、大学院生など様々なバックグラウンドの方が受講していました。
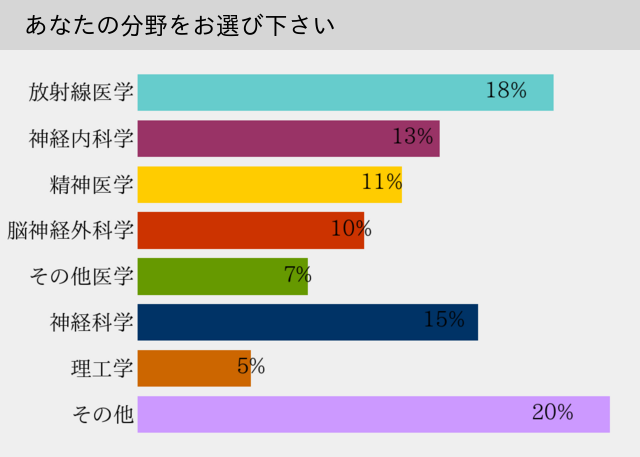
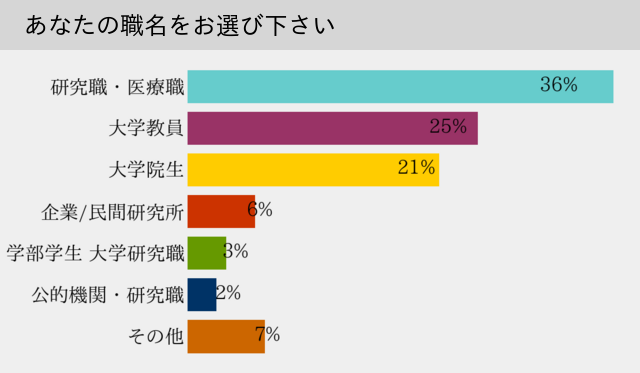 放射線医学、神経内科学、精神医学、脳神経外科学など様々なバックグラウンドの方が受講していました。
放射線医学、神経内科学、精神医学、脳神経外科学など様々なバックグラウンドの方が受講していました。
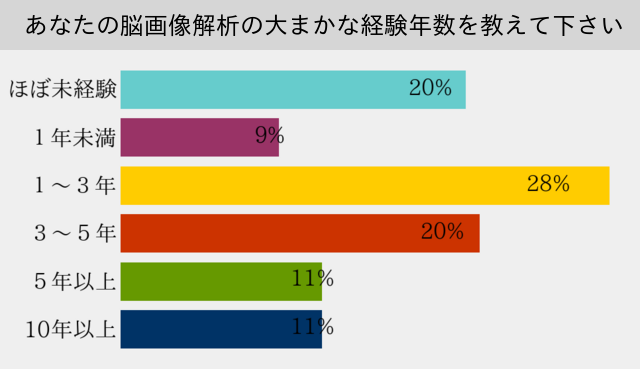
 57%の受講者が脳画像解析は未経験または経験3年未満でした。
57%の受講者が脳画像解析は未経験または経験3年未満でした。
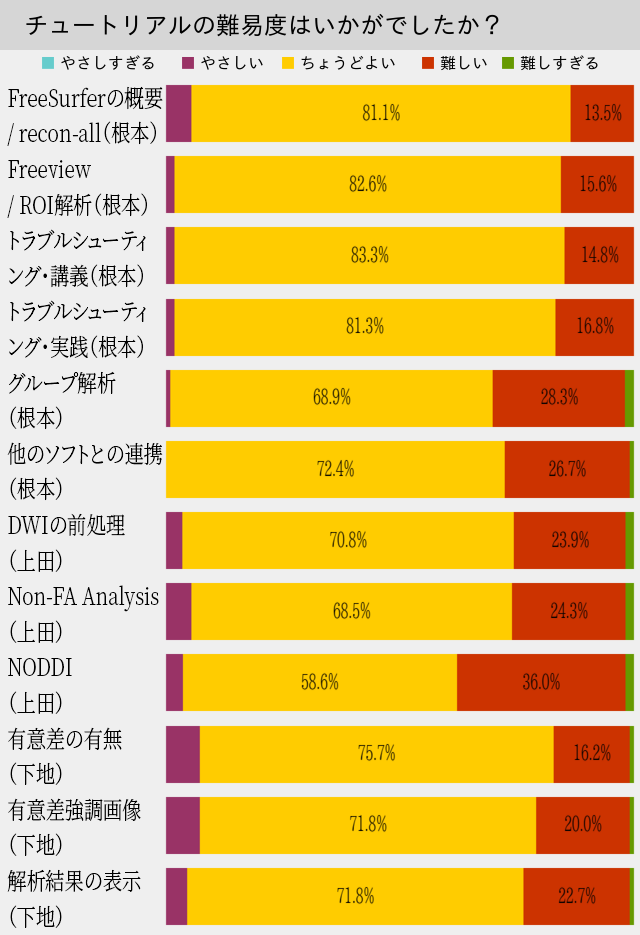
 58~83%がちょうど良い難易度と回答しています。
58~83%がちょうど良い難易度と回答しています。
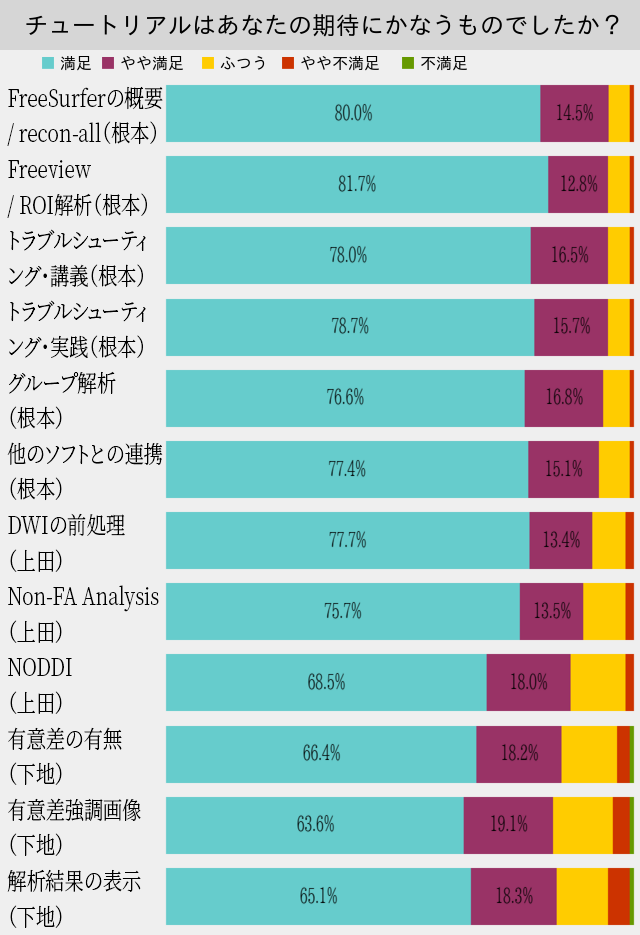
 82~94%が満足~やや満足と回答しています。
82~94%が満足~やや満足と回答しています。
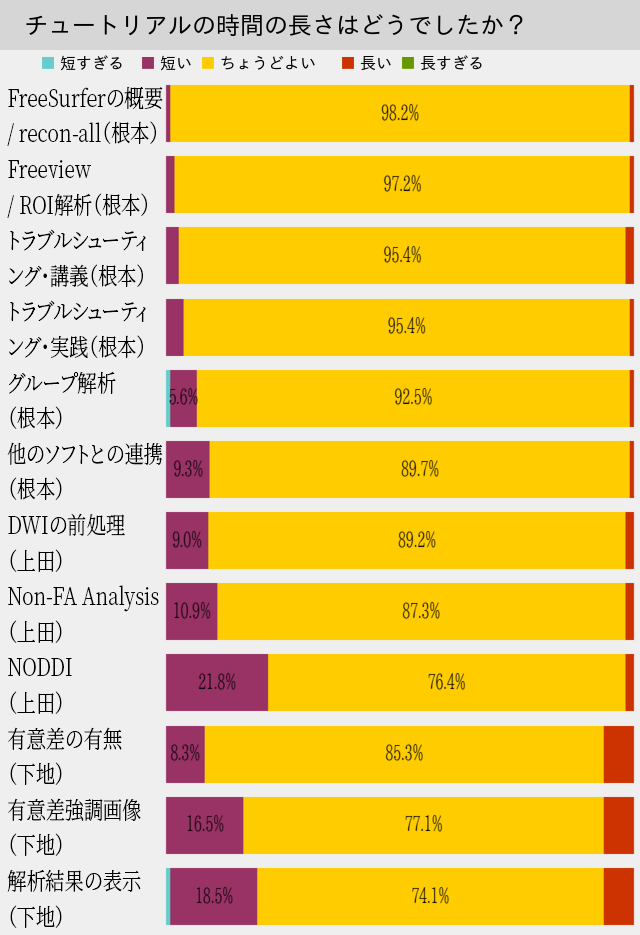
 74~98%がちょうどよい長さと回答しています。
74~98%がちょうどよい長さと回答しています。
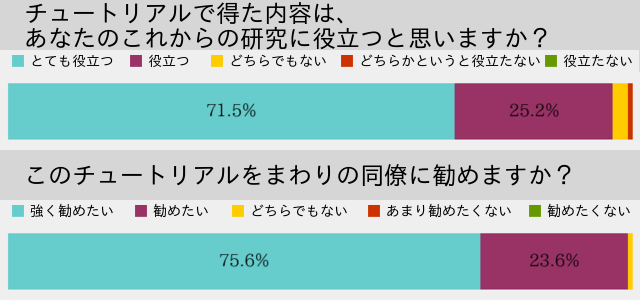
 受講後アンケートでは96%がこれからの研究にとても役立つ、または役立つと回答しています。
受講後アンケートでは96%がこれからの研究にとても役立つ、または役立つと回答しています。
今回のチュートリアルで良かった点をご自由にお書きください。(抜粋)
- 1月に行った基礎的な部分を復習するタイミングもあり、より実践的なチュートリアルで非常に勉強になりました。
- DTIの画像変換を含めて全体的な流れが理解できた。
- DTIの結果の扱い方なども大変参考になりました。
- DTI講義の中でこれまでになかったNODDIの講義を聞けたことが大変有意義でした。
- DWI解析が非常にわかりやすく有用だった。
- FreeSuferについてはだいぶ理解が深まった。
- Freesuferの基礎やscriptについて前回の内容をさらにわかりやすく学ぶことができました。
- FreeSurferのトラブルシューティング(実習)がとても良かった。
- FreeSurferのトラブルシューティングの行い方を教えていただけた点が良かったです。
- FreeSurferの概要や新しい内容のアップデートを聞けたことが有意義でした。
- FreeSurferの概略と利用方法などが理解できた。
- FreeSurferの実際の使い方をFreeviewや画像変換を含めてご教授頂けて勉強になりました。有難うございました。
- FreeSurferは使用したことがあり,recon-allも使用経験がありました.しかし,概要をあまり理解しないままに使用していたため,基本的事項を確認できてよかったです.
- FreeSurferは初心者のため、概要からはじめてくださり分かりやすかったです
- FreeSurferやTBSSなど自分は関与していない領域のことだったので難しかったが、少し全体像が分かった。
- FreeSurferを独学で学ぶのは非常に難しいので、先生の講演を受講する機会を頂けて大変勉強になりました。
- FreeSurfer全体を詳細に解説していただき、全体が把握しやすく詳細に解析しやすいと感じました。
- Jupyter Notebookが使いやすかった
- linuxコマンドについても先月の復習も兼ねていただけたので理解が深まりました。
- NODDIなど新しい解析を学べた
- NODDIについて全く知らなかったのですが、非常に勉強になりました。生理学的な解釈が与えやすいということもあり、ぜひ今後の研究に活かせるように復習します。
- NODDIについて知ることができた。
- NODDIの話が聞けて良かったです。2shellのデータを取得できる機会があれば、是非解析してみたいです。
- Pumbleでいつでも質問できる雰囲気が良かったです。
- Pumbleになってあとから確認しやすくなった。他の人の質問も勉強になる。
- Pumbleのスレッドを増やしていただいているおかげで幅広い質問が出しやすくなったと感じました。
- Pumbleへの質問にもすぐに回答してくださり、疑問がすぐに解決したのでよかったです。
- Pumbleもとても有機的で、受講して本当に良かったと思います。
- Pumble素晴らしいと思います。別の機会に活用したいと思います。
- scriptに関しては慣れが必要と感じた(文法なので)。
- synthSRやinfantFSなど興味のあった点に触れて頂き、また、トラブルシューティングについてもわかりやすく解説頂き、大変勉強になりました。
- いずれの講義でも、基本となるコマンドラインの意味を細かく丁寧に読み解いて頂いたのは大変助かりました。
- いずれの講義も勉強になりました。今回はNODDIについても時間を取って扱いがあり大変勉強になりました。
- いつもながら基本的なところから解説していただけてありがたかったです。
- いつものように丁寧に解説して頂き,質問にも詳しく回答して頂きました
- おかげさまでFreeSurferの流れが系統だって理解できた。
- オンデマンドで見返すようにして下さっているので、復習ができて非常にありがたいです。
- グループ解析は,今後の研究で応用してみたいと思います.
- コピー&ペーストで追いつけました。事前データが処理されていて追いつくことが出来てよかったです。
- コマンドを書けるようになるのは言語学習と同じように忍耐が必要だと感じました。
- これまでと比べて時間に余裕があり、より理解しやすく受講することが出来ました。
- これまでの受講者からの質問を紹介いただき,よくあるような質問を回避できた.
- これまでの断片的な知識が繋がりました。ありがとうございました。
- スクリプトの詳細を知れたこと。
- スクリプトの内容について繰り返し説明していただいたので、理解が深まりました。
- スタッフの方々の丁寧な対応に感激でした。
- チートシートを使用して、コピペでスムーズに実施できました。
- チュートリアルの録画や、履歴が消えないPumbleの利用など、どうしてもリアルタイムには100%吸収できないことを復習したりする機会があること。
- テキストが充実していた
- テキストが大変わかりやすく、予習や復習にとても役立ちます。
- どういうときにどのような解析を使うのかが分かってとても良かったです。
- とても丁寧に詳細に教えてくれた点
- どの講義も丁寧で、とても学習しやすかったです。
- トラクトグラフィーなどの違いなどについて概説してくださっており、自身の理解の確認にとても役立ちました。
- トラブルシューティングの方法を学ぶことができてよかったです.
- 以前からの内容に新しいことを加えていただき、毎回勉強になります。
- 演習と合わせて行ったため、実際の操作方法についても理解が深まったので、今後の研究に生かせると思いました。
- 下地先生が1月の内容の復習を入れてくださっていた点。
- 下地先生のスクリプトの威力が凄まじかったです。どうもありがとうございます。
- 解析の手順について丁寧に説明していただき、理解がすすみました。
- 解説はとても丁寧で分かり易かったです。
- 各処理の説明をしていただいた後に実践できたので、より理解しやすかったです。丁寧に説明していただけて良かったです。
- 拡散強調画像も基礎的な部分から理解できた。
- 基礎的な内容も解説なさってくださって助かります。
- 基本事項からひとつひとつ丁寧に教えていただき,知識の整理ができました.本当にありがとうございます.
- 休憩時間を確保されたので助かりました。
- 休憩時間を十分に取ってくださり、集中力を保ちやすかったです。
- 教材のコマンド部分のコピーが簡単にできるのが良かったです。ご準備いただきましてありがとうございました。
- 広い視野で教えていただけた点
- 講義内容のバランスがとても良かったと思いました。後半は時間が足りず早くなって、受講者がついていけなくなるということが無いようにご配慮いただいたと思いました。
- 今回のコースの下準備として1月に入門編があったことがとても助かりました。ありがとうございました。
- 今回のチュートリアルで良かった点をご自由にお書き下さい
- 今回のチュートリアルをしっかり復習して、解析対象を具体的にし、改めて研究を進めて行きたいと思いました。貴重な機会をいただきありがとうございました。
- 根本先生のrecon-allの解説がとても勉強になりました。
- 根本先生の講義は、自分で解析をしている時にどうしたらいいのかわからないようなことを取り上げていただいており、実践的でわかりやすくとても勉強になりました。
- 最近拡散MRIについて学習を始めたので、その復習や新しい発見ができた点が非常に良かったです。
- 昨年のチュートリアルをベースに、復習とさらなる技術の取得に役立った
- 資料がとてもまとまっていて素晴らしいです。
- 資料が豊富だった
- 資料が豊富なので自己学習としてスライドを使用できそうなのも嬉しかった。
- 資料も充実しているため後から見直すことで理解を深めることができました。
- 事前に募集していた疑問点などを講義の中で書面で残してくださっているので非常に参考になりました。
- 自身の研究で使用してみた後に改めて聞くと、見え方が変わると思ったので、今後に活かしていきたい。
- 自分は初心者なので概要を知ることが出来て良かったです。まずは自分の症例でもやってみたいと思えたので良かったです。
- 質問に対する対応も充実していました。
- 重要なポイントを丁寧に説明して頂いたことが大変良かった。非常に参考になりました。
- 初心者の自分では難しく感じる部分も多かったですが、非常に丁寧にご講義頂いたのでしっかり復習しようと思います。
- 書籍等では分からない解析方法を取り上げていただいたこと
- 上田先生のチュートリアル時に前臨床データを使用する場合に注意すべきポイントに言及いただいたことが勉強になりました.
- 上田先生の拡散の講義で、JupyterNotebookに記載されていた今までの質問とそれに対する回答が、みなが聞きたい(と思われる)内容が集約されていてよかった
- 新しい知見が身についた
- 数年前に受講した際よりも資料や講義内容がブラッシュアップされ、理解しやすくなっていた。
- 先生方の丁寧な講義により、全くわからない所から、なんとなく出来るかも!?と思うようになりました。
- 専門的な知識がないため、わからないところも多かったが、脳画像解析についての見聞を深めることができた。
- 前回からmac nativeで参加しております。前回は自身のPCに異なるバージョンのpythonが多数インストールされており、コントロールするのに苦労しましたが、今回はその方法についてもご教示いただきとても助かりました。ありがとうございました。
- 前回と比較して、基本の点をしっかり触れられていて、わかりやすい講義でした。下地先生の講義は、今回わかりやすく、コマンドを触れてみようという人が増える気がしました。
- 前回のアンケートに記載した内容を取り上げていただきとても感謝しております。
- 全て勉強になりましたが、特に上田先生のNODDIの解説が自分のやりたいことに近くて特に勉強になりました。
- 相当量の濃い内容を圧縮してご教示いただいたと考えております。研究の雰囲気が少しですが、理解できたように思います。感謝しております。
- 他のソフトウェアとの連携ついて,大変参考になった.
- 多大な情報量にも関わらず、jupyter notebook やチートシートの活用によって受講者が置いていかれることなく進んでおり、大変満足度の高い内容でした。
- 大変分かりやすく勉強になった。
- 大変勉強になりました。来月もよろしくお願いします。
- 長年やってきても知らなかったオプションや、理解せずに入れていたeddyオプションに関する理解が深まった
- 内容がよく練られていました。
- 難しい内容をテキストやPumble、解説で大変わかりやすくご教示下さり、大変感謝申し上げます。
- 豊富なサンプルを用いて、実際に動かすことができて楽しかった。
- 未経験でしたが、先月の下地先生のコマンド入門を受けていたので、DTI解析の講義内容に比較的ついていくことができました。
- 両日とも新しい知識を学べました。
- 論文や解析ソフトのウェブサイトを読んだだけではよくわからないが、実際に解析し論文を書くまでに必要なTipsをたくさん、かつ、わかりやすく説明してくださり、非常に勉強になった。
次回のチュートリアルに向けて改善できる点がありましたらご自由にお書き下さい。(抜粋)
- DWIも大変貴重なテキストを(ただでいいのか?と思います)頂いているので、説明部分と実践部分をある程度まとめられてもいいかもと思いました。
- FreeSurfer統計は難しいと思いました。理解と実践を同時に行うと、一瞬の聞き漏らしが致命的に感じてしまいがちです。遅れたらコピペでいいのですが、その部分の理解に穴が開くと、その後はコピペを続けるだけになるので、ある程度座学で居場所を理解したほうが良いように思いました。
- GLMの解析について詳しく知りたい(Design matrixの作り方など)
- Jupyter notebookを久々に使ったため,コピペのやり方に戸惑い,一度デフォルトのスクリプトを弄ってしまい自分で初期化して遅れてしまいました。Jupyter notebookの基本的な操作方法を教えて頂けたらより嬉しかったです。
- Linux実環境でのセットアップ(NODDIやGPUの実践など)、Linux実環境で参加する人向けの資材提供
- Zoomだと聞き逃しを確認するのが難しいです。Youtube Liveのように時間を少し戻ったり追っかけができる環境だと嬉しいです。後日動画をアップロードしていただいていますので、大きな問題ではないです。
- あとからパッと見直す際にPDF資料が最初からあると大変助かります
- サブマスク画像とは、という部分、時間を割いて丁寧に解説いただいていたのですが、(わたしが)文字の理解が少し苦手なため、視覚的にどういうものなのかイメージがあるとよりどんな作業をしているのかイメージしやすいかもしれないと感じました。
- ジュピターノートブックは慣れていないので、ターミナルを使用した方が分かりやすかった印象をもちました。
- ソフトの初回起動時は少しだけ時間をとっていただけるとありがたかったかもしれません。
- ダウンロード資料のファイル名を名づけのルールが講師の先生方の間で統一されているとありがたいなと感じました。
- とくにありません.
- 一つの講義の時間がやや長い(私の集中力の問題とも言える)
- スクリプトの内容を、それぞれ詳しく教えてほしかったです。
- 各コマンドの解説より各スクリプトの具体的な使い方をもう少し聞きたかったです
- 拡散MRIの話は理解に時間がかかってしまっていたので、もう少しゆっくり時間をかけて説明していただけたらありがたいです。
- 教材PDFがどこにあるのか、ぱっと見わかりづらかったです。
- 講義ごとに休憩を入れてもらっていますが、1日を通じて午後からの講義の集中力が低下している感覚があります。午前中や午後だけなど1日の長さを短く日数を増やすなどして頂けると個人的に有難いです。
- 今後も基本的なところを抑えつつ、新しい情報を盛り込んでいただけたらと思います。
- 今年度は6日間に分けられていて事前に十分に予定が組めます、大変に感謝です。
- 細かいLinuxコマンドレベルのお話と研究の高水準の画像処理のお話があり個人的には若干迷子になりました。
- 時間配分
- 時間配分の関係もあるのだとは思いますが、下地先生のコマンドの話をもう少しゆっくり聞きたかったです。
- 自分のデータでやるとなったときにまだ少しハードルを感じます。そのあたりへの注意点やポイントがもっと増えるといいなと思いました。Pumbleでだいぶ解決できそうでもあります。
- 十分に練られていると感じました。
- 初心者のため作業に気を取られて内容を聞き逃す場合があるので,こまめにテキストの該当箇所等をリマインドしていただけるとついていきやすくなるかと思いました.
- 統計解析手法についてもう少しじっくりと勉強できればと思いました。
- 特にありません
- 特にありません。
- 特にありません。とても頑張っていただいていると感謝しています。
- 特にありません。大変膨大な内容をまとめていただきありがとうございました。
- 特にございません。
- 特にないです。
- 特になし
- 特になし。
- 特に改善していただきたい点はありません。
今回のチュートリアルでとりあげられなかった内容で今後とりあげて欲しい内容はありますか?(抜粋)
- 「TIPS:diffusion MRIがどのような研究でどのように使われているのか」で様々な解析法の特徴を整理してくださっていましたが、そこをもう少し詳しく掘り下げる時間を設けていただきたかったです。
- ANTsについて講義していただきたい。
- command_line内の各スクリプトの実践的な使い方をぜひご講義ください。
- Disconnectome Symptoms Discovererのような、新しい解析技術の概要も取り上げてほしい
- DKI、CSDなどdMRIの様々な解析
- DTIトラクトグラフィー(TRACULAなど)
- DWI解析で他のモデル(DKI等)も取り扱ってくださると幸いです。
- FBAについて
- FBSS
- free water DWIなど新た手法についても今後勉強させて頂ければと存じます。
- FreeSurferの縦断解析
- Googleや生成系AIを用いたscriptの作成方法や画像データ取得に関することをより深く教えていただけたらと思います。
- GQIなどについても触れていただけると理解がより深まると思いました。
- NODDIの具体的導入(インストール)やAMICOのインストールなど
- PET画像の解析に興味があります.PET-surferというソフトウェアがあるようですが,その使用方法について勉強してみたいです.
- TBSS以外のDTI解析
- tractographyの描出方法の講義があれば非常に嬉しいです。
- TRACULA等のトラクトグラフィー
- Voxel-based lesion symptom mappingを学んでみたい。
- workbenchのソフトの使用法を学びたいです。
- ここで聞いた内容を軸に、伸ばせる人は伸ばすと思いますので、十分と思います。
- トラクトグラフィー
- トラクトグラフィーの解析
- トラクトグラフィの作成について
- マスク画像(インデックスマスクやサブマスク)について十分理解できなかったので、復習したいと思います。
- 安静時fMRI解析・TaskfMRI解析における機能的結合性の解析
- 応用としてfMRIprepなどのパイプライン解析を扱っていただければと思います。
- 画像ビューワーの表示設定をコマンドで指定することが多かったのですが、GUIの場合の操作方法についても触れていただけたら嬉しいです。
- 解析手法の選択には、撮像に費やせる時間と目的による、ということは理解しておりますが、どのような「目的」の時には、どれがベターなのかをもう少し知りたいと思っております。
- 拡散強調画像におけるtract specific analysis (TRACULAなど)についても、時間をさいていただけばと思います。
- 構造画像のコネクティビティ解析
- 講義や質問で時折触れられていたDKIの内容もぜひ扱っていただきたいです。
- 今回は処理の仕方が主であったように感じましたのでDTI等に関してもう少し相関など実例での解析の仕方などを教えて頂けるとありがたいです。
- 今回取り上げられなかったスクリプトについて、以前のテキスト等を共有して頂けるようでしたらありがたいです。
- 撮像プロトコルにおける注意点やポイントについて
- 撮像条件と解析結果の関係、アーチファクト対策など撮像に関する内容があると嬉しいです。
- 施設でいかに画像を取得するかだと感じましたので、チュータの皆様のご意見や参加されているご施設の状況など解析環境についても知りたいです。
- 次回1日目だけが仕事でどうしても参加できません。またビデオをアップロードしていただけたら大変有り難いです。
- 縦断解析についてご教示いただけますと大変うれしいです
- 新しい解析手法の解説や比較をしてくださると嬉しく思います。それは論文を読む上でも有益かと思いました。"
- 深層学習や機械学習など
- 前臨床データを使用する場合に,チュートリアル内容から変更すべきポイント等に言及いただけると,より直接的に研究内容に役立てることができるため希望いたします.
- 大変満足できる内容で、講師をはじめとする先生方にとても感謝しております。実際にfreesurferを解析に取り入れたいと思います。その際、疑問に思う点等ありましたらぜひご教授いただきたいと思っております。
- 脳血流シンチグラフィーの解析(SPM)
- 脳波、MEGの解析(SPM)
- 勉強を始めたてのため、他にどのような内容があるのか理解できていません。その内容にどのようなものがあるのかだけ教えていただけると講義に後に自分で調べることもできて良いと思います。
- 論文で使われているデータの統計分析をトレースするような実習があるといいなと思いました。
- 論文に乗せる図表のスタンダードな設定とその方法
その他、ご意見ありましたら自由にご記入下さい。(抜粋)
- 1時間ごとくらいに休憩がとれて、集中力が保てました
- 2回目の参加でしたが前回よりよくわかりました。繰り返し勉強する機会をいただいて大変感謝しております。どうもありがとうございました。
- Freesurferも拡散強調画像もすぐに使う予定があったわけではありませんでしたが、大変勉強になりました。自分でいじるようになったら改めて参加したいと思います。
- Pumbleの質疑応答も参考になるものがたくさんあり、ありがたいです。
- Pumbleの質疑応答も利用しやすく、チューターの先生のおかげもあり、普段から疑問になっている事を整理することが出来ました。大変貴重な講習会、ありがとうございました。
- Pumbleも来年度は書き換えでしょうか。このまま継続できれば大変うれしいです。
- ありがとうございました
- ありがとうございました。"
- いつもありがとうございます。3月もよろしくお願いします。
- いつもありがとうございます。講師の皆様、チュータの熱意に毎回感動いたします。ぜひ今後も続けていただければと思います。
- お忙しいところ、大変有意義な勉強会を開いていただき、ありがとうございます。今後も可能でしたら、継続して受講したいです。
- この2日間、本当にありがとうございました。所用で2日目の午後は参加できませんでしたが、いろいろな知識を吸収できました。ありがとうございました。
- このようなハイクオリティな講義を無料で受けさせていただけることに感激しております。ありがとうございます。
- この講習会が無料というのはすごいことだと思います。ありがとうございます。
- これまで何回か受講していますが、いつも新しい情報提供に驚かされます。
- しっかり復習して自分の研究に役立てられるようにしたいと考えています.
- スタッフの皆様に感謝しかありません。ありがとうございました。
- テキスト等やデータ等の準備も膨大な時間を要したことと思います。先生方が一生懸命研究された内容を惜しみなく、しかも私のような初心者にも大変わかりやすく教えていただき本当に感謝申し上げます。
- もう連続して4年ぐらい参加しておりますが、毎回新しい発見があります。運営スタイルは毎回更新されていますし、内容は基本は踏襲しつつも、毎回新しいことが含まれています。自身の技術レベルの向上を感じます。ありがとうございます。
- 安静時、taskのfMRI解析楽しみにしています。
- 一日がかりの講習会ですが,毎回充実していて,時間があっという間に過ぎます.とても素晴らしい講習会に参加できて,大変光栄です.
- 引き続き、研究に役立つチュートリアルが存続し、発展して行くことを願っております。本当にありがとうございました。またよろしくお願い致します。
- 下地先生が丁寧にコマンド演習をしてくださるので、コマンドはあまり経験がなかったのですが苦手意識が少しずつ薄れてきました。講義をありがとうございました。
- 回を重ねるたびにチュートリアルが、わかりやすくなっていると思います。
- 貴重な情報や経験を共有いただけますこと心より感謝しております。自身の研究成果に活かせますよう頑張る所存でおります。今後ともどうぞよろしくお願いいたします。
- 勤務と重なってしまったため、全ての講義に参加出来なかったのが残念です。次回ありましたらまた参加させて頂きたいです。
- 講師の先生方、2日間、本当にありがとうございました。熱量が伝わってきて、今後の研究のアイデアやモチベーションアップにもつながりました。
- 講師の先生方には多くの勉強をさせて頂き、大変感謝しております。
- 今回の講義も大変勉強になりました。特にNODDI解析を知らなかったので、ぜひ挑戦してみたいなと感じました。
- 今回も大変勉強になりました。講義のおかげで画像解析ソフトでどのようなことができるかがわかってきたので、今後研究で困った時に解決法を見つけやすくなるのがありがたいです。
- 根本先生をはじめご準備いただきました皆様、ありがとうございました。大変勉強になりました。
- 準備ならびに講義やチューターで参加頂いた先生方に、深く御礼申し上げます。ありがとうございました。
- 専門的な知識がなく、医師でもありませんが、とても有意義な時間を過ごすことができました。ありがとうございました。
- 大変系統だったチュートリアルで勉強になりました.また事前を含めてPumbleでの手厚いサポートも非常にありがたく存じます.
- 大変勉強になりました。ありがとうございました。次回もよろしくお願いします。
- 知識を整理するのに、非常によい機会となりました。どうもありがとうございました。
- 丁度手元のデータでDWIk解析をやってみよう、と思っていた所です。チュートリアル拝聴して「これならイケそう。」と感じました。3月もお世話になります。
- 非常に参考になるご講義をありがとうございました。研究が前進できそうです。
- 普段はMATLAB,SPM環境においてGUIで解析していましたが,段々とlinux環境のFSL・FreeSurfer等をシェルスクリプトで動かすことに慣れてきました。無償ソフトを用いて画像解析できるようになれると今後自分の力になるだろうと一層思いました。引き続き勉強させて頂きます。
- 復習を繰り返し行いたいので、今回も動画配信をお願い致します。
- 分からないこともチャットで質問を受け付けて下さりこちらの疑問を解決することができました。このような勉強する機会を頂けて感謝しております。
- 本当にありがとうございました
- 本当に膨大な内容をわかりやすくまとめていただきありがとうございました。
- 毎回、丁寧に対応して頂いて本当にありがとうございます。講師の先生方、チューターの方々に感謝です。
- 毎回1回では覚えきれないほどの膨大な情報量で演習についていくだけで精一杯です。しっかり復習したいと思います。次回もとても楽しみです。ありががとうございました。
- 予定が入ってしまっており、全日程の参加ができないのですが、後日ビデオ配信があり大変助かっております。お忙しい中至れり尽くせりの対応をしていただき、感謝申し上げます。
- 来月も勉強させてもらえればと思います。
2024年2月ABiS脳画像解析チュートリアル
【日 時】 2024年2月17日(土)、2月18日(日)【会 場】 完全オンライン(Zoom、Pumble)
【参 加】 事前登録制(参加費無料、PC環境が準備できない場合は受講できないことがあります)
【概 要】
2月17日(土) FreeSurferチュートリアル
08:30 Zoomオープン
09:00-09:20 開会、オリエンテーション
09:20-10:20 (1) FreeSurferの概要 / recon-all (筑波大・根本)
10:30-11:30 (2) Freeview / ROI解析 (根本)
11:40-12:10 (3) トラブルシューティング・講義 (根本)
12:10-13:10 昼休み
13:10-14:20 (4) Freeviewを用いたトラブルシューティング・実践 (根本)
14:40-15:50 (5) グループ解析 (根本)
16:00-17:00 (6) 他のソフトとの連携 (根本)
17:00-17:30 質疑応答
2月18日(日) 拡散MRIチュートリアル
08:30 Zoomオープン
09:00-09:10 開会、オリエンテーション
09:10-10:10 (1) DWIの前処理 (慶応大学病院・上田)
10:20-11:20 (2) TBSS (Non-FA Analysisも含む) (上田)
11:30-12:30 (3) NODDI (上田)
12:30-13:30 昼休み
13:30-14:30 (4) TBSS解析後の有意差の有無 (順天堂大・下地)
14:40-15:40 (5) TBSS解析後の有意差を強調する画像 (下地)
16:00-17:00 (6) TBSS解析後の解析結果の表示 (下地)
17:00-17:30 質疑応答
開催済みの脳画像解析チュートリアル
2024年1月20日~21日 2024年1月ABiS脳画像解析チュートリアルを開催致しました。
2023年1月28日~29日 2023年1月ABiS脳画像解析チュートリアルを開催致しました。
2022年12月11日、17日~18日 2022年12月ABiS脳画像解析チュートリアルを開催致しました。
2022年1月29日~30日 第11回ABiS脳画像解析チュートリアルを開催致しました。
2021年12月18日~19日 第10回ABiS脳画像解析チュートリアルを開催致しました。
2021年1月30日~31日 第9回ABiS脳画像解析チュートリアルを開催致しました。
2020年12月19日~20日 第8回ABiS脳画像解析チュートリアルを開催致しました。
2020年2月8日~9日 第7回ABiS脳画像解析チュートリアルを開催致しました。
2019年3月2日~3日 第6回ABiS脳画像解析チュートリアルを開催致しました。
2018年12月22日~23日 第5回ABiS脳画像解析チュートリアルを開催致しました。
2018年3月3日~4日 第4回ABiS脳画像解析チュートリアルを開催致しました。
2018年1月20日~21日 第3回ABiS脳画像解析チュートリアルを開催致しました。