MRI研究の裾野を広げていくことを目的に脳MRI解析チュートリアルを開催します。チュートリアルでは十分な解析手法を持たない国内各地の研究者を対象に、実際に脳MRI解析を体験してもらいながら実践的な脳MRI解析手法を実演、説明を行います。
2022年12月ABiS脳画像解析チュートリアル
2022年12月11日(日)、17日(土)~18日(日)の3日間、完全オンラインで「2022年12月ABiS脳画像解析チュートリアル」を開催致しました。
チュートリアルは盛況のうちに終了致しました。多数のご参加ありがとうございました。
大学院生、研究職・医療職、大学教員など様々なバックグラウンドの方が受講していました。
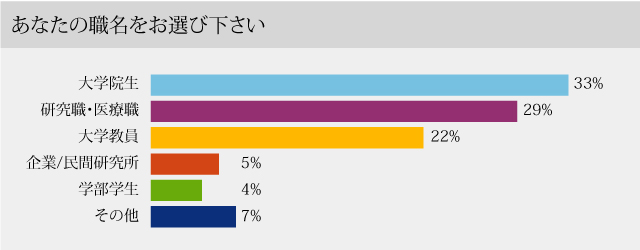 放射線医学、神経内科学、精神医学など様々なバックグラウンドの方が受講していました。
放射線医学、神経内科学、精神医学など様々なバックグラウンドの方が受講していました。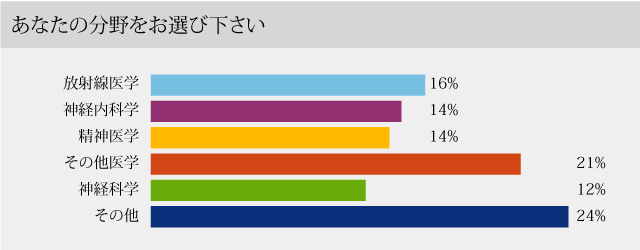 76%の受講者が脳画像解析は未経験または経験3年未満でした。
76%の受講者が脳画像解析は未経験または経験3年未満でした。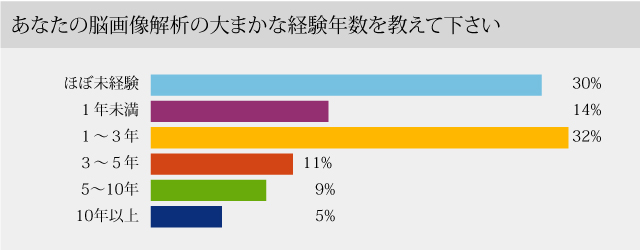 33~83%がちょうど良い難易度と回答しています。
33~83%がちょうど良い難易度と回答しています。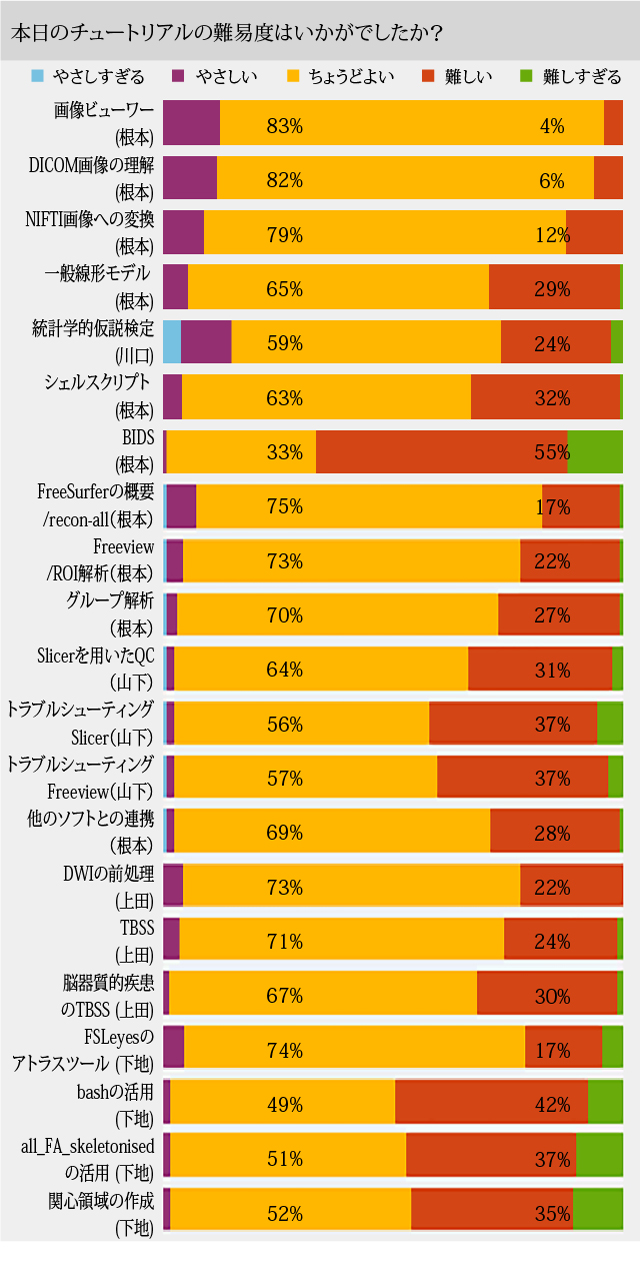 76~96%が満足~やや満足と回答しています。
76~96%が満足~やや満足と回答しています。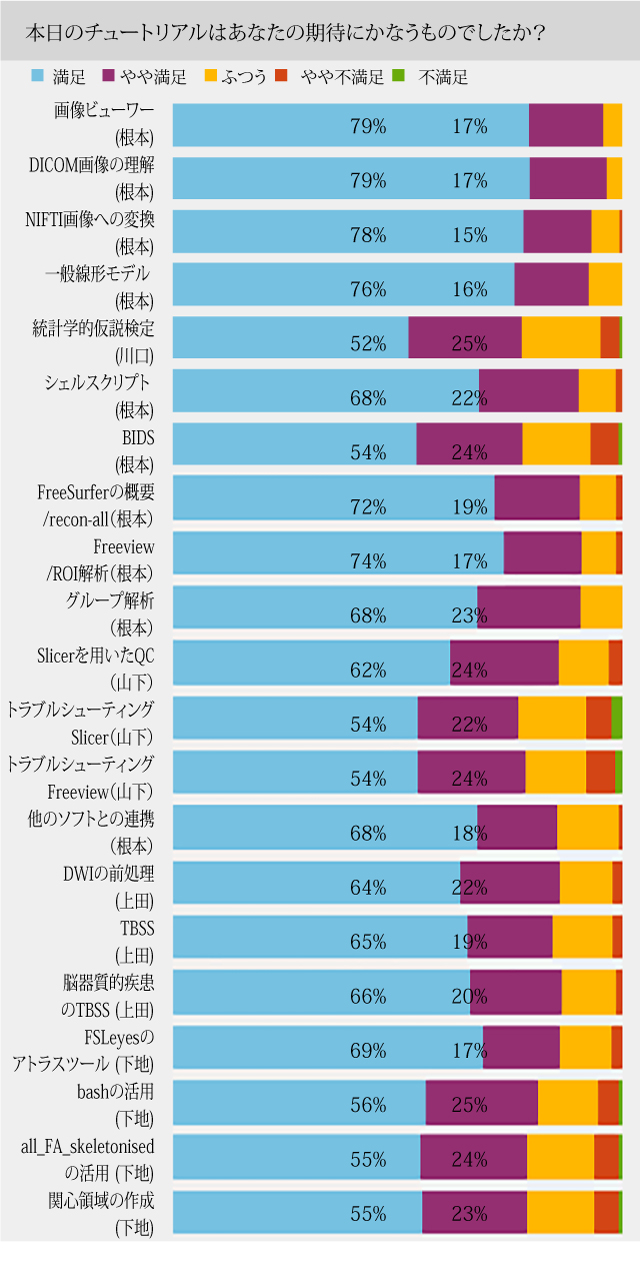 70~97%がちょうどよい長さと回答しています。
70~97%がちょうどよい長さと回答しています。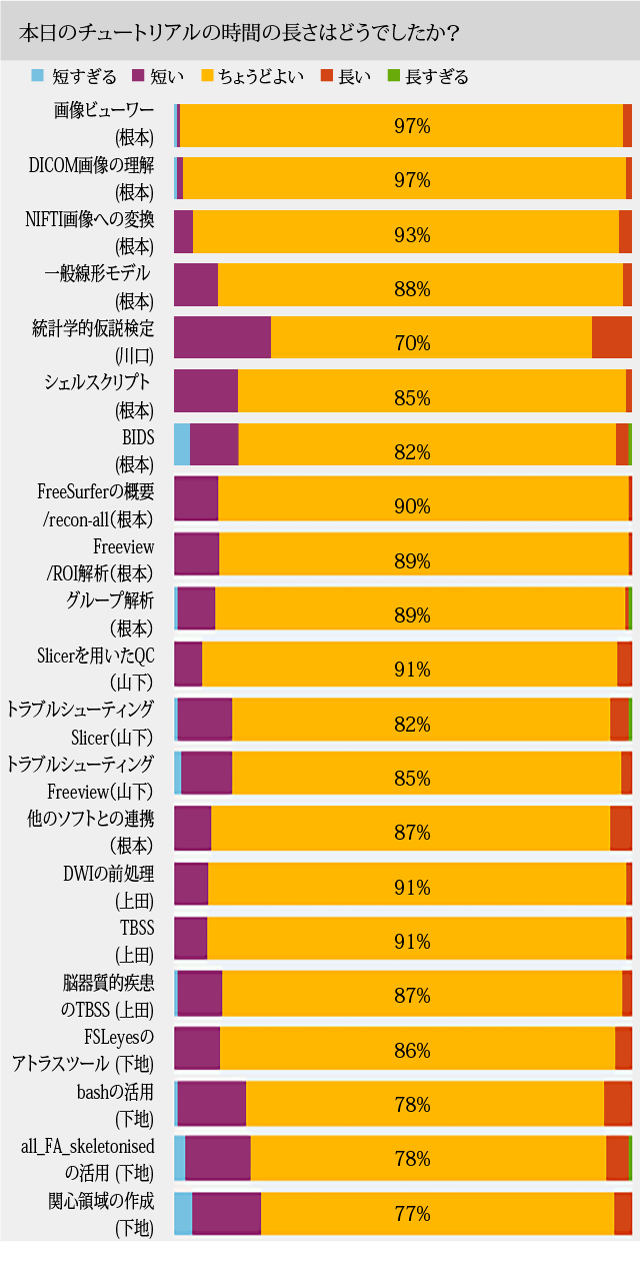 受講後アンケートでは98%がこれからの研究にとても役立つ、または役立つと回答しています。
受講後アンケートでは98%がこれからの研究にとても役立つ、または役立つと回答しています。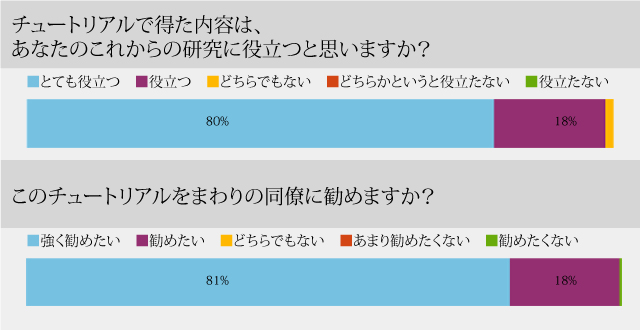
今回のチュートリアルで良かった点をご自由にお書きください。(抜粋)
- あとから動画で復習できるのがいい。
- いつもながら丁寧にご指導いただき大変ありがとうございました。
- いつもわかりやすい講義ありがとうございます。
- オンラインでの講義でしたが、サポート体制が事前段階からとても手厚く、大変助かりました。
- オンラインで実施していただいたことがよかったです。
- オンラインのため自宅で受講できた。
- コピーシステムや適宜ターミナルに打ち込むめりはりがあって勉強になりました。
- コピーボタンが大変わかりやすかった。
- コマンドコピー便利でした。
- コマンドのコピペは大変便利でした。
- コマンドをコピーして使えたのは非常に良かったです。
- コマンド入力の面白さが少し感じられたのが特に良かったです。
- これまでよりも各セクションの時間が短く設定されていて、集中して講習を受けることができました。
- これまで通り、非常によく準備されたチュートリアルでした
- サポートの先生方のご対応のおかげで大変勉強になりました。
- シェルスクリプトでのtipsがとても役に立ちそうです。これまで手作業でやっていたものを自動化でき時間の短縮かつエラーも軽減できそうでありがたいです。
- シェルスクリプトの使い方やターミナルベースの操作は、独学でなかなか理解できずとっつきにくかったのですが、やはり1から示してくれる人がいると、理解がぐっと進むもんだと感じました。
- スクリプトなど慣れないことを知るよいきっかけになりました。
- スクリプトをコピーできるようにしてくださっていた点が大変よかったです。
- スクリプト処理の紹介
- すべての講義において、丁寧な説明とテストデータでの模擬実践ができてよかった。
- すべての内容がわかりやすく、大変勉強になりました。
- スラックで気軽に質問できたところ
- ソフトウェアでの解析だけでなく,解析法に関する周辺情報やbashのscript解説,またデータの保管のされ方などまで教えて頂けた点が良かったです.
- ターミナルに入力するコマンドの意味を適宜説明しながら講義を進めていただき、この操作はなにをどうしているのかを理解しやすかったです。脳画像解析の知識があれば、いろいろ応用が効くんだろうなぁと思いながら拝聴しておりました。
- ターミナルに入力する作業が増えていて、体で覚えられた感じがしました。
- ターミナルの入力はまだ難しいですが、非常に面白かったです
- ターミナルの利用
- チートシートが便利だった
- チートシートは,ミスタイプの心配もなく,先生方のスピードについていくことができたため,大変使用しやすかったです.
- チートテキストがユーザーフレンドリーに改善されており,スムーズに講義についていくことができました.
- チューターの先生方、お疲れ様でした。ご丁寧にありがとうございました。
- チューターの方々がすぐに対応してくださったので多少つまづいても挽回できたのでよかったです。
- チュートリアル、テキストの通りにやれば、一通り動かすことができ、正解がわかるので、今後、自学する際に今日のことがヒントになり活用できると思いました。
- ちょうどFSLeyesでROIを作ろうとしていたところだったのでとても参考になった
- ちょうど解析で困っているところが口頭のコメントで聞くことができて有難かったです。
- データの取り扱いやFigureの作成など、より具体的になっていること。
- テキストが初心者にもよりわかりやすくなっているように感じました。復習する際に大変助かると思います
- テキストが丁寧に作られていたので、初心者でもテキストと先生の説明を聞きながらでなんとなく理解ができました。
- テキストだけではなく生のお話を聞けるところが勉強になります。
- とてもよくまとまっていて感動しました
- とても統計に関して説明がわかりやすかった.
- とにかくテキストが丁寧でわかりやすいです。
- どの単元もすっきりした構成と明確な目標で、今後の研究に直ぐに活かせます。
- どんな質問にも丁寧に答えてくださる点です。
- バックアップがとても手厚い
- ビデオがあるのであとで復習しやすいこと
- ブラックボックスだったrecon-allの中が知れてよかったです。
- プログラミングの経験が無いに等しい状態で参加させて頂きましたが,全体の流れなどが洗練されており,スムーズに導入でき,解析の特徴などをイメージすることができました.
- ほかの方の質問や先生方の回答から勉強することも多く,大変貴重な時間となりました.
- ほぼ未経験ですが、基本的なところから順番にご講義いただいたので、とてもわかりやすかったです。
- また何に対しても例がついていたので、見ていてやってみて実感できる点が良かったです。
- ゆっくりでついていきやすく感じました
- レスポンスが非常に早くわかりやすかった
- わかりやすかったのと概要が分かるのが助かりました。
- 以前のアンケートで希望したことをいれていただいていたので有難かったです。
- 一つ一つのセクションで学びが多く、大変勉強になりました。
- 一般線形モデルの理解が曖昧だったので、今回の説明で非常に理解することが出来ました。
- 音声が聞き取り易く、ストレス無く拝聴できました。
- 下地先生の「seq -w 100 |」はすごくわかりやすかったです。パイプの概念、よく理解できていなかったのですが、すごくわかりました。書き写すコマンドの文字数が少ない、結果表示が簡単に理解できる、ことによると思います。パイプ、すばらしいですね!
- 何となく使用していたスクリプトについて理解することができた。
- 何度かFreeSurferやFSLの内容を聞いていたのですが、小技などがちりばめられていて、研究に応用することが出来ると感じました。
- 画像データハントリングの基礎を学べて大変良かったです。
- 画像の取り扱いについて基本から教えて頂いた点
- 画像解析の基礎を学べたのがよかったです。
- 画像解析の基本的なところも含めて解説してもらえた。
- 画面を共有していただきながら解析を進める事が出来たため,分かりやすかったです.
- 会期中のSlackによるスタッフの皆さんのバクアップがありがたかったです。
- 解析結果で有意差の出るところがたくさんあったりするときの考え方など分かって良かったです。
- 皆さま丁寧なご説明を有難うございました。
- 各ソフトが何をしているものかなど把握することができた。
- 各項目に十分な時間を取ってもらえたこと、拡散MRIで器質的疾患の作業内容、根本先生の他のソフトとの連携、論文化を視野に入れた立ち位置での作業の仕方についてご教授いただけたことが特によかったです。
- 拡散MRIの基礎的な概説や測定法のバリエーションの話があったので,今回の講義内容の位置づけが分かりやすかった
- 慣れている先生がターミナルへコマンド入力する速度を体感でき、とても勉強になりました。
- 関心領域の作成も非常に臨床的に有用だった。
- 器質的損傷を伴う解析はとても有意義だった。
- 基礎の基礎から解説してくださり大変わかりやすかったです。
- 基礎を教えて頂けたことです。
- 基礎的なことを学ぶ機会がなかったので、大変勉強になりました。ありがとうございました。
- 基礎的なことを丁寧に教えて頂けて本当によかった。
- 基礎的なところでありながら、あやふやなところをしっかり勉強出来ました。
- 基礎的な内容が非常に重要かと思いますので、大変有意義でした。
- 基本的だが今まで教えてもらえなかった内容が含まれていた。
- 基本的なソフトの使い方の基礎から勉強になりよかった。
- 基本的なところから、引っかかる点までフォローが行き届いていて大変参考になりました。
- 基本的な統計の話をじっくり聞くことができて、個人的には良かったです。
- 基本的な部分から丁寧にお教えいただき大変勉強になりました。
- 疑問点についてもSlackでスタッフの方々が、素早く丁寧にお答えくださり、円滑に勉強することができました。
- 休憩時間があって助かりました.
- 去年の講習会も参加させていただきましたが,最新の情報が含まれていたり,macのネイティブ環境でも受講できるように対応されていて,講師の先生方,チューターの先生方がお忙しい中たくさんの時間と労力をかけて準備してくださっていることを改めて感じました。
- 結果表示の例を示していただいたこと。
- 現在NIFTI形式で画像解析をしてますが、BIDS形式へはすぐに移行しないつもりですが、常識として抑えることができて感謝しております。
- 現在解析で必要な情報を得られたこと。
- 後日,受講できなかった内容をビデオなどで復習できること
- 講義と事前の資料がすばらしかったので、迷うことがなかったです。
- 講義の速度についていけない箇所があったため,動画が残っているのが大変ありがたいです。
- 講義を聞きながら追えたので理解しやすかった。
- 講義資料がわかりやすかったです。ジュピターも使用しやすかったです。
- 講師が講義を進めてくださり、チューターの先生方がSlackの質問に回答をしてくださり、万全のサポート体制で講義を受けることが出来てありがたかった。
- 講師の先生の資料がとても分かりやすかったです。
- 講習会中にもっと良い方法について質問が出たり、他のチューターの先生からのご発言もあり、解析はこうして試行錯誤の中でよりよい方法を見つけていくものなのだということを実感できた点。
- 今までよりもチューターの先生方のresponseの速さと回答の完成度の高さに驚きました。
- 今まで作業でやっていたことなど、全体に見通しが立った
- 今回習得したことをベースに、自分で独学していけそうな気がしてきました。
- 今後のチュートリアルも大変楽しみにしています。
- 根本先生のls, cdの速さ
- 根本先生のご講義はいつものごとく素晴らしく、自分の扱っているデータの意味を改めて理解することができました。
- 作業の進め方だけでなく、一般線形モデルや脳画像統計のお話も聞けて非常に勉強になりました。
- 資料・録画もあるので、復習がしやすい点がよかったです
- 事前にテキストやデータを受け取り,当日に講師の先生が実際にデモを行って頂き,同時に自分自身のPCで実際に行うことができますので,とても有りがたいです.
- 事前に資料を早めに配布してくださり、予習時間を十分にとってくださった点。
- 事前のトラブルシューティングの案内があり、当日混乱を避けられた
- 事前の予習とセットアップのサポート、当日の丁寧なサポート、事後学習の整備などすでてにおいて感謝しかないです。
- 事前質問で、チュートリアルまでの間も勉強させていただきました。
- 事前質問に答えてる。
- 事前準備からそのサポートを含め、非常にわかりやすかったです。
- 事前準備のサポートから講義内容まで非常に充実していたところ。
- 事前準備の仕組みを準備されていて、とてもわかりやすかったです。
- 事前準備の段階から実際の講義まで、サポート体制がとても充実していたと思います。
- 自分にとっては難しい内容も基本的なことから非常に分かりやすく説明いただき勉強になりました。有難うございました。
- 自分のPCで参加できる点で、受講後も自分で試してみることができ、有難いです。
- 自分はあんまり慣れていない内容もありましたが、根本先生の細かい説明のおかげで自分の中で”なるほど、次からこれ使いたい”と何度も思いました。手作業よりもコードでできることが良いなーと。
- 質問しやすい環境
- 質問におけるチューターの先生方の迅速な対応をいただき大変ありがたく思います。
- 質問にも丁寧にお答えくださり、ありがとうございました。
- 質問に回答してもらえたこと。
- 質問に対して即座にご対応いただけたのでよかった。
- 質問に逐一答えてくださるのもありがたかったです。
- 実際にターミナルへの入力作業があったので、自分の中で整理し、理解しながら受講できました。
- 実際に使用しているコマンド入力の意味などよく理解できました。
- 実際に自分で解析をしながら、講義を受けることが出来た点が非常に良かったです。
- 実際の流れを自分でスクリプトを触りながら体験できたことも、自身での研究などのとっかかりに非常に優れたものだった。
- 実際の論文の方法をトレースしていくのはよかったと思う。
- 実践的でわかりやすく、参加してよかったです。
- 実践的な内容で、コードの意味も非常に丁寧に解説して下さったので、自分のデータでも同様の解析ができそうです。
- 手を動かしながら行うところ、概念的なところの組み合わせの構成が良かったです
- 手厚く講義いただきありがとうございます。
- 受講者が気になったことをすぐSlackで質問できる空気ができていると感じました。
- 受講者の皆様からの質問に、その場で答えてはるのを、聞けたことがよかったです。
- 準備が素晴らしく、多少遅れても取り戻せることができる安心感が素晴らしいです。
- 準備万端して、説明優しい、理解やすい
- 初めての参加で全てを一度に理解することは難しかったので、復習用に動画を後でみかえせれるようにしてくださった点
- 初学者から中級者へ行く過程で、復習と未知の知識の二つを知る事ができた。
- 初学者が入りやすいように、話の構成や順序がしっかり構築されていて大変ためになりました。
- 初学者だったので、基本的なことがハンズオンで勉強できて大変よかったです。
- 初学者にも分かりやすく教えていただけた点、最新の知識・技術について(理解が及ばないまでも)教えていただけた点
- 初参加だったが、事前の環境準備など含めて、内容についていけたため良かった。
- 初心者ですが、ちょうど良いペースで講義が進んだので、理解が深まりました。ありがとうございました。
- 初心者でもすでにあるコマンドを実行するだけで進められたので遅れずに進められました。
- 初心者でもできるようにしてくれている。サポート体制が整っている。オンデマンドがある。
- 初心者でもわかりやすい
- 初心者で不安があったのですが、基本中の基本から教えて下さったので、全くついていけないということにはならず、とても勉強になりました。
- 初心者に向けた丁寧なご講義をありがとうございました。本当にありがとうございました。参加してよかったです。
- 初心者に向けでスクリプトを打つ時間を作っていただいたりうまくいかない時にどこから修正したらいいか説明していただけて分かりやすかったです。
- 数回連続で参加させて頂いておりますが、毎回新しい話題に触れて頂き、かつ論文作成にも通じる知識を提供いただいており(今回MRIcroGLやBIDS形式のことはとても新鮮に伺いました)、たいへん興味深く聴講いたしました。
- 先生の説明が分かりやすい
- 川口先生のご講義は非常に非常にわかりやすく、ヒトに説明する時にはこのようにしたらよい、というお手本を学ばせていただいた気がします。時間がなく途中スキップしたクラスターとボクセルのお話も、ぜひ聞きたかったなと思いました。
- 線形代数や統計についてもさらに深く学ぶ必要があるなと感じました。
- 前回TBSSのチュートリアルを受講しましたが、今回はその復習ができ、さらには器質疾患がある場合やFA値のROI解析までご教示頂くことができ大変勉強になりました。
- 前回のチュートリアルからさらに新しい情報量が増え、聞きたかった部分の詳しい解説が聞けて大変勉強になりました。
- 前回もそうだったが資料や環境がきちんと準備されていること。
- 全体的に大変勉強になりました。
- 素早いチューターの方々のご対応に感謝です
- 大変有意義でした。
- 大変有意義で今回も受講してよかったと感じています。
- 知識のアップデートができるので、勉強になります
- 中々の他では学べない分野に関して先生方が非常に詳細に深く解説くださり大変勉強になりました。
- 途中でもSlackでサポートしていただけたので有り難かったです。
- 当日の内容もさることながら,事前準備,講義および講義資料,チューターの方々の対応,いずれも満足のいくものでした.
- 統計について詳しく解説いただけた点
- 統計やGLMなど、自学できにくい事項が学習できた。
- 統計学のところは苦手であったため、雰囲気を感じることができました。ありがとうございました。
- 特にシェルスクリプトの部分で丁寧に説明していただけて非常に勉強になりました。苦手意識が減ってスクリプトを扱うのが楽しくなりそうです。
- 内容が豊富で多岐にわたっていること、新しい情報が取り入れられていること、実際の研究や論文作成に使用できる内容が盛り込まれていることがとても良かったです。
- 内容的にちょうど3日間で行なって、楽に参加ができました。
- 日本語で受講できる点.ハンズオンで理解しながら進められる点.
- 入門編の方への指導の仕方について、とても勉強になりました。
- 脳画像解析の基礎をしっかり学習できた
- 脳画像解析の経験はほぼ未経験だったので、初日に初心者のためのチュートリアルを開催していただけてとてもよかったです。
- 脳画像解析の専門家から,初めて体系的に解析の基礎を学べたこと.今まで独学で解析を行ってきたが,その内容が客観的に正しいのかどうかを知ることができた.
- 脳画像解析はほぼ未経験だったため、今回の講義を理解できるか、ついていけるかとても不安でしたが、大変丁寧に教えていただき、理解することができました。ありがとうございました!
- 脳画像解析は初心者で,コマンドを使用するのも不慣れであったため不安がありましたが,丁寧なご説明とガイドラインだったため,大変理解しやすくついていくことができました。
- 脳画像解析初心者のためついていけるか心配でしたが、非常に丁寧に解説していただいてよくわかりました。
- 脳解析初心者でしたが、丁寧に解説いただいてなんとかついていくことができました。
- 脳病変がある場合の解析など、昨年までのチュートリアルになかった内容を追加していただいた点がとてもうれしく思います。
- 非常に丁寧、実践的で、充実した内容でした。質問にも一つずづ答えていただけるのはありがたいです。
- 非常に勉強になりました.gitやSlackを活用したトレーニングは初でしたが,非常に便利な講義スタイルだと思いました.
- 普段扱う機会のないFreeSurferや3D Slicerについて詳しく学べた点と,TBSS解析においてGUI・CUI両面での結果の出し方が学べた点が非常にありがたかったです.
- 普段気づかない点や,最新のトレンドなどを理解することができ,大変勉強になりました.
- 復習できる点、terminalを用いてCUI操作に全くなじみがなかったが復習して練習したくなりました。
- 復習もしやすいようにしていただいているので、しっかり勉強して今後に生かしていきたいと思います。
- 復習用の動画がとてもありがたいです。
- 復習用の動画の公開は大変ありがたいです。
- 複数回きいていますが、皆様進化しておられて、いつも勉強になります
- 毎回、同じテーマでも内容が進化していて、受講するたびに知識が増えるのと、解析経験が少しできてきてから受講した今回は、自分の手持ちデータの解析を考えながら受講できたので、とても勉強になりました。前回、前々回の講義が、点と点で繋がっていくような感じがしました。
- 毎回思っていますが、事前準備・会期中のサポート・講演内容、など、良いことしか思い当たりません。
- 毎回情報が更新されていてとても勉強になります.
- 毎回新しい知識を仕入れることができ,本当に勉強になります!ありがとうございました.
- 毎回内容が更新されていて,今回は根本先生のBIDSの話と川口先生の話が特に勉強になりました.
- 問題が起こった時に先生たちは素早く返信して助かります。
- 用語として聞いたことはあっても細かいところまで理解できていなかった部分や解析の全体の流れ等ご説明いただけたのでよかった。
- 録画ビデオなどで復習しやすくして頂いているのありがたいです。
- 録画もあり,いつでも復習できる環境であることがありがたい.いつでも復習ができ,身につきやすい
- 録画を公開していただけるのもありがたいです。録画をみながら復習しようと思います。
- 論文の方法に沿った講義なども理解しやすかったです。
- 1から説明してくださったところが良かったです。
- 1箇所つまづいた点がありましたが、後から動画を共有してくださるとのことで大変助かります。
- 2回目ですが、丁寧回答してくださって感謝しております。
- 3度目の参加でしたので,前回よりは明らかに理解が進みました.
- 3日間になったことで、FreeSurfer、TBSSの内容に踏み込んだチュートリアルになっており、ありがたかったです。
- ABiSのチュートリアルに参加し始めてから2年か3年かほどですが、ほぼ毎回講義で取り扱う内容に変化があり、普段通りの研究生活を送っていたら知らなかったことなんだろうなと思うような新しい知識のチュートリアルを体験することができていて、今回もまた新しいことを実際に手を動かしながら体験することができたので、そこがよかったと思う。
- AlizaMS、Surf-iceは使用したことがなかったのですが、わかりやすく明日からすぐに使えそうな情報でした。
- AlizaMSは知らなかったので勉強になりました。
- Bashの活用について、以前より根本先生のスクリプトを参考に、画像解析と別の仕事のファイル処理に使わせて頂いておりました。今回のチュートリアルでも新たに参考になる情報がありました。
- BIDS化は苦労していたので、今回の講義で共有していただいたスクリプトをもとに実際に自分でもやってみたいと思います。
- DICOMデータの取り扱いで認識不足を埋めて頂きました。
- DICOMについては復習できました。
- DICOMの扱い,変換について復習が出来た点とBIDSに関して新しい知識が得られた点。
- DICOMの扱いに関して詳しくしれたこと(これまであまり重視していなかった)。
- DICOMファイルの変換、ソートで新しい知見を得ることができた。
- DTI画像のTBSS解析では、結果の画像が得られたものの、そこからどのように実際にそのskeleton画像を使用したらよいのか、イメージができていなかったので、大変勉強になりました。
- For構文、while構文の解説は画像解析などは大助かりなのでとてもありがたいです。
- FreeSuferというソフトのことが理解できた点、DWI前処理で新しい情報を得られた点がよかったです。
- FreeSurfer, DTI画像の解析ともに、実際に使用している最中だったので、理解が深まりました。
- FreeSurfer, DWIの解析ともに未経験で、はじめての内容ばかりでしたが、概要をつかむことができました。
- FreeSurfer, MRtrix3, FSLといったソフトの奥深さがわかった。
- FreeSurferについて日本語で丁寧な解説をうかがえたのがよかったです。
- FreeSurferのQC、DTIのアトラスの活用法やROI解析周りなど、個人的にうまく独学できずにいた内容を多数ご講義いただけて大変ありがたかったです。
- FreeSurferのrecona-allと、どのようにrecona-allが成されているか、またうまくいかないときの対応方法を学ぶことができました。
- FreeSurferの解析に関して,学びを深くすることができました.日頃の解析で悩む点がクリアになりました.
- FreeSurferの基礎からしっかりと学ぶことができた点
- FreeSurferも、recon-allをしてみたことはあったものの、複数症例を一気に処理したり、トラブルシューティングなど、実際的な使い方がわかっていなかったので、大変勉強になりました。
- FreeSurfer解析の概要がわかったこと。
- git機能の利用は簡単・便利でよかったです。
- GLMについて、コントラストを立てる意味がとてもよく理解できました。
- GLMの解説、BIDS化の講義がとても参考になりました。
- GLMの基礎的知識の整理とDICOM画像やその変換、コマンドやスクリプトなどについて一通り学ぶことができた点。
- jupyter notebook でマークダウンを追える点。
- jupyter notebookの使用によって流れを追いやすかった。
- jupyter notebookの利用が大変便利でした。
- jupyter-notebookでの実例がとても分かりやすかったです。
- Jupyternotebookの利用でコマンド入力せずにスムーズに進めた
- lin4neuroの存在を知ったことが大きかったです。macで解析をしているのですが、色々とインストールが煩雑でしたので、一括でまとめられるプラットフォームがあるとは知りませんでした。
- recon-allのトラブルシューティングやDTIのROI作成など知りたかったテーマをうかがえたのがよかったです。
- Slackでの質問、応答の経過を読んで後からでも見ることが出来るのが良いです。
- Slackでの質問はし易かった。
- Slackでほかの参加者でも生じた問題などを確認しながら進めたので,大きな遅れがなく講義についていけてよかった.
- SlackとZoomを使用した新しい講義形態でしたが、手元が見やすい、細かい対応をしていただける、他の方の質問も見えるなど、メリットが盛りだくさんで本当に満足でした。
- Slackの利用がよかったです。準備資料も読みやすかったです。
- Slackを使用して、チューターの方に質問をしながら進められるのはとてもよかったです。
- Slack導入がすごくよかったです。
- TBSSで脳解剖学的多様性のある方のデータを処理する際のノウハウが学べたのが収穫でした.
- TBSSに興味があり、MRTrix とfslの内容が非常に勉強になった。
- TBSSの活用を検討するいい機会になりました。
- TBSSの追加解析について、ちょうどやりたい内容だったので有り難かったです。
- TBSSをちょうどしていたので上田先生の講義が大変勉強になりました。
- TBSS解析の流れがわかったので、初心者としては良かったです。
- TBSS解析後の追加解析の意義を教えていただけて良かったです。論文を読むときにも、今までより細部に気を付けて読めそうな気になりました。
- TBSS後の解析について知識が深まった
- Unixの使い方で勉強になることがたくさんありました。
次回のチュートリアルに向けて改善できる点がありましたらご自由にお書き下さい。(抜粋)
- うまくいかなかった時に先に進んでしまうので、さらについていけなくなる
- オンサイトとオフサイトの両方のメリットがあるので今後はハイブリッドを希望します。
- オンラインでできる限りのことをしていただいたと思っております。
- クラスター補正の部分が一番知りたい部分でして、他の研究者で議論が盛んなポイントだと思いますので、次回のチュートリアルでは是非詳しいご説明をいただけましたら幸いです。
- ございません.素晴らしいかったです.
- このような内容で十分です。
- コマンドの意味についてもう少し説明があると初心者でもわかりやすいと感じた.
- コマンドを打つときの十字キーでショートカットできる機能をはじめの方に教えていただきたかったです.
- コマンド情報などまとめのチャンネルをSlack上で作っていただけたら、と思いました。
- コマンド中心の講義では、コマンド入力に慣れていないため、意味を理解するのにだいぶ時間がかかるので、ゆっくり解説して欲しいです。
- これまでABiSで取り扱ってこなかった発展的な解析についても触れていただきたい
- さらに具体例が豊富であると良いです。
- ジュビターノートブックよりは、チートシートでコピーペーストの方が直感的でわかりやすく感じました。
- スクリプトについてのトレーニングについてもう少し内容を増やして欲しかったです.
- スクリプトになると突然難しく感じてついていけなかった。
- スクリプトに不慣れなため、予習してから参加させていただきたいと思います。
- スクリプトを打つのは勉強になるのですが、初心者のためスクリプトを打っている途中で先生の画面の解析が始まりコマンドが見えなくなり、何を打ったら良いのか分からなくなってしまう点が困りました。
- スケジュールが時間単位でほしいと感じました。
- スケジュールどおり(ほぼ進んでいましたが)に進めてもらえると集中力が保たれやすいので,改善いただけますと有り難いです.
- ターミナルにタイプする文字(打ち込む文字)は多くあるため,意味を覚える方法がありましたら,ご教授いただきたく存じます.
- ターミナルにタイプする文字の意味の理解があいまいなまま進めてしまっていたところもありました.
- ターミナルのコピペボタンがあると,エラーやミスタイプがあったときにも講義に追いつくことができると思いました。
- ターミナル打ち込みですが、私が不慣れであり講師の方が、windowsであるので適宜操作がことなっている部分を補うことが大変でした。パスがうまく通りときはいいのですが、うまくいかなかったときに修正ができず置いていかれてしまいました。
- タイトなスケジュールなので難しいとは思いますが、参加者同志の交流が持てるような時間がとれると良いですね。
- タイプミスなどで手間取り講義についていけなくなる可能性もあるのですが、個人的には以前のようなチートシートからターミナルへコピペする形式も捨てがたいなとは思いました。
- チートシートはjupyterになっているものも全セッションあるとうれしいです
- データの更新があった際のSlackでの連絡などを引き続きピン留めして欲しい
- データをダウンロードしそびれていたため、講義の最初で躓いてしまった。講義に必要なデータの事前チェックリストやチェックシステムがあると良いと思った。
- テキストのどこの内容を話しているのかがわかるように示していただけると助かります。
- テキストブックで予習していたのですが、17、18日は内容がヘビーだったため、テキストブックのアップデートをもう1日だけ早くしていただけましたら嬉しかったです。
- どうしてもM1 Macだとエラーになってしまうことが多く、やりづらいところもあった。
- とくにありません。
- とくにございません
- トラクトグラフィーも取り入れていただきたかったです。
- ないです。
- ないです。
- ないんじゃないでしょうか。
- なし
- ノートパソコンを利用してマウスを使っていないことも、すこし考慮して進めていただきたかったです。特にviewerの利用はGUIベースなので、その点が苦痛でした。
- パイプ、すごく良いのですが、パイプの理解は、パイプ前のコマンドが理解できていないとつまづきます。
- プレゼンのスビートがちょっと遅くなってほしい
- まったくございません。
- マルチシェルの場合にシングルシェルとは違ってどの部分の解析に気をつけたらいいのか、今回のスクリプトはシングルシェルにしか使えないのか、それともマルチシェルでも可能なのか、そのあたりがわかりにくかったです。
- 一度エラーなどで遅れを取ると話についていくのが少々困難でした。
- 音質
- 何度か参加していることもあり、スクリプトがうまく通らない時や途中で止まってしまう際の対処法が自分で見出せるようになってきました(Windowsです)。
- 画面上の操作を一瞬でも見逃すとテキストでキャッチアップすることも難しいと感じました。
- 解説時間が足りなくなってしまったようで、もう少し解説に時間をかけて欲しかったところがありました。
- 改善点を探すのが難しいです.
- 開始時刻をもう少し遅くしていただけると助かります.
- 休み時間で先生が質問に答えてくださりすぐに次の講義が始まるので、先生が休憩されずに大丈夫か心配になりました。
- 休憩時間をもう少し長くとっていただけると嬉しいです。
- 休日3日連続よりは、土曜日3回や半日に分割するなどしていただけるとスケジュールを組みやすいと感じた。
- 結果表示の際に欠かせない数値DATAなど査読者目線でご教示いただきたい。
- 研究への応用例(実例)などをご紹介頂けると,3D slicerの魅力がよりわかりやすくなると感じました.
- 見ているだけではなかなかコマンドが覚えられなかったので、少しだけでも小テストのように自分でコマンドを書くセッションがあればよいと感じました。
- 講義でいくつかのプロセスに分け、解析頂いたが、実際の流れみたいなものを知らないので、自分にとっては各プロセスが断片的で、これをどのような流れで解析までつながるかのイメージが繋がりにくいと思いました。
- 講義資料pdfはweb上ですとテキストがコピーできないので打ち込みに苦労しました。
- 講義内でなくてもよいと思いますが、演習や課題があると習得できたかを確認できると思います。
- 今のままで素晴らしいと思う内容です。
- 今回はグループ解析まで一歩進んで学ぶことができましたが、次回はもっともっとグループ解析をつっこんでやりたいです。
- 今回は技術的なことに重きをおかれていたと思います。脳画像解析が歴史的にどのように発展して、各技術がどのような研究でどのように使われるのか、今後の技術の方向性などについても簡単なガイダンスが最初にあると、学ぶ意義が深まるように思います。
- 資料の中で誤りがあった部分は後ほど差し替えていただけますと幸いです.
- 事前準備を行っていたが、初学者のためついていくことが難しかったです。
- 種々の論文を例に作図方法をさらに教えてほしい
- 受講者の一つ一つの作業の間の時間をもう少しとってほしかった講義があった。
- 周知事項が埋もれて操作に手間取ってしまうことがありました。
- 十分なサポートで感謝しております。
- 初学者なので、後半はずいぶん難しかったです
- 初心者には最後の3コマ(下地先生のコマ)は,途中で脱落するとどうしようもない状況に陥ってしまいましたが,中級者以上の方にはとても良い内容だと思いました.
- 少しスピードが速く、つまづいた時に追いつくのが大変だったが、オンデマンドで復習できるのは助かります。
- 正直、改善点はない位大変感激しました。
- 盛りだくさんの講義なのでじっくり5日間くらいかけて学びたいです。そのくらいボリュームが多く、素晴らしかったです。
- 説明内容について概念的には理解できるが,どのように自分の研究に適用できるのか,各項目における応用例を示してもらえるとよりよかったように思う
- 川口先生の統計のお話しをもっと時間をかけて聞きたかったです.最後に少しされたクラスター推測の話など,特に初学者の方には勉強になる部分かと思いました.
- 全体的に初学者を想定されていることを承知の上で,統計検定に関しては少し基礎的すぎたように感じました。もちろん重要で,昨日の内容が不確かな研究者が少なからずいるもの確かですが,もう少し最後の方の,クラスターレベル補正等の脳画像解析に特化した内容をお聞きしたいと個人的には感じました。
- 素晴らしい講義の中で画像への統計解析に対する解説が駆け足になってしまい、そこを主に聴きたかったので残念でした。
- 素晴らしい内容で、改善点は特にありません。
- 早く受講を締め切られていた点。次回も受講したい為
- 大きくはありません。
- 追加資料や初学者用のテキストなど手厚くしてもらえると助かります。
- 土曜日は午後からだと助かります。
- 統計についての講義は時間が短く、後半は駆け足で難しく感じました。じっくりと話を聞ければ、なおよかったと思いました。
- 統計のところが、後半を聞きたかったが、駆け足でさらっと終わってしまったので残念でした。
- 統計の初心者としては数式の話というよりはグラフとp値がどういう関係になっているかなどを詳しく講義していただければ幸いです。
- 統計解析は、最初の入りがややわかりにくかったですが、FWE, FDWがとてもよくわかりました。逆にその時間が短かったのが残念です。
- 特にありません。
- 特にありません。ありがとうございました。
- 特にありません。素晴らしかったです
- 特にありませんが、少しスピードが早い点があり、見逃し・聞き逃したときに10秒戻る・・・などできたら便利だなあと思いました。
- 特にありませんが先生方やスタッフの負担が心配です。
- 特にございません。
- 特にございませんでした。
- 特にない
- 特にないです。
- 特になし。
- 特に無いです。
- 難易度が高く、初心者では途中からついていけませんでした。
- 難易度を教えていただけると嬉しいです(初心者向けの内容など)
- 日程とのバランスもあるのかと思いますが、半日だと、楽だなと思いました。
- 脳画像統計は最初の部分は短くてもいいので、もう少し画像特有の統計の話が聞きたかったです。
- 半日程度のコースだと参加しやすいです。
- 予習をもっとしておけばよかったと後悔しました。
- 予習教材があったことに直前まで気づいていなかったので,もう少し強くアナウンスしていただけたほうがよかったです。
- 理想論ですが、週末に2回よりは、3週に渡って1回ずつの方が復習しやすいと感じました。
- 連日開催だと疲れるように思いました。
- ~/git/fs-scriptへのパスを通しておらずちょっと慌てました
- 12/17,18の講義後半は、駆け足になりがちで話が頭の中で追いつかなかった。内容を減らすか時間の調整をお願いしたい。
- 1日通して行うのが、なかなか大変でした。
- 1日通しは夕方になると疲れてくるので、半日程度で複数日に分けて行う方法はありますか?
- 3D slicerについて,基本操作は事前学習を必須とした方が学習効率が上がる気がしました.
- 3日目の演習では,自分でターミナルに入力するほうが勉強になると感じたため,保険としてコピペボタンがあるとありがたいと思いました。
- ABiSのホームページの情報更新が遅く受講のチャンスを逃すなと思いました。
- fileコマンドとシェルスクリプト上のファイル変数fileが、初学者に混同される恐れがあるので、ファイル変数名にfileは使わない方がよろしいかと思います。
- GUIベースの解析時にはどのボタンを押すのか迷子になることが多かったので,テキストに全て記載いただけると助かります。
- jupyter notebookよりもターミナルで自分の手を動かして進めていく方が、理解が深まった気がします。
- jupytar notebookでつまる所が多かったので、初心者にさらに易しくご指導いただければ幸いです。
- jupyter notebookは大変便利と思いますが、ターミナルへ直接入力する経験があったほうが、チュートリアル後に実際に自分で解析をしようとする際のイメージがしやすいと思いました。
- jupyter notebookにも書き込み、メモが自由にできると利用しやすい
- Lin4Neuro以外の環境で処理を行う場面がある場合、気持ちゆっくりめに講義していただけますと、初学者としては助かります。
- Linuxのスクリプトになれていない身としては、Codingの際に説明していただく内容をフォローすることで精いっぱいで、なかなか全体の構造の中で自分が今何をしているのかを把握するのが困難でした。
- MacOS nativeあるいはMacのキーボードだと違う操作が必要な点が多々あり、講義に追いつくのが難しいことがありました。
- MacOS nativeでのエラーが生じることがありました。
- macOSで参加したので、一部講義通りに進まず、スタックして追いつけないことがあった。
- Macの方々はいろいろと大変そうだなという印象でした。OS問題は本当に大変ですね。改善点ということではなく、Macのエラーに対処する方法が見出されるといいなあと思いました。
- MRI解析の操作中心のことを期待していたので統計の基礎のようなものは、自分には不要であった。FDRなどの考え方は重要であるが、そのような知識的なものは他にも学習の機会があると思うのでオプショナルなものであった方が良かった。
- NIFTIへの変換などについても、ゴールまでの流れを示していただいたうえで、このコードが全体のどの部分にあたるのかなどがわかるとなおさら理解が進むように思われました。
- PDFファイルのテキストで画像になっているものは、ファイル内で検索できないので、なるべくテキスト情報が残る形で提供して欲しいです。
- Slackでトラブル報告を"トラブル報告"など別のチャンネルに書き分けることで見返しやすくなるかな、と感じました。動作トラブルなどが質問チャンネルに混ざると若干見づらくなるようにも感じました。
- VirtualPC以外にLin4Neuroを動かせる環境のご検討(parallelsなどM1Mac向けに)
今回のチュートリアルでとりあげられなかった内容で今後とりあげて欲しい内容はありますか?(抜粋)
- クラウドの運用とセキュリティ
- グループ解析、縦断解析に特化した講義があると、とても嬉しいです。
- げっ歯類等の非臨床MRI画像の解析に関して,処理や可視化の点で臨床画像と異なる部分があるのか知りたい
- コネクトーム解析について
- シェルスクリプトの使い方をより詳しく知ることができる講座があれば是非受講したいです。
- それぞれの脳MRI解析でFSLやFreeSurfer, MRtrix3など専用の解析環境がある中で、脳MRI解析でのMATLABの活用方法(どういうシチュエーションでmatlabを使用しているか)について、様々なTipsも含めて知りたいです。
- ターミナルについてもう少し詳細を勉強したいです。
- ターミナルの基本について。bash、zprofileなど種類があって、その使い分けや、Macにおいてどう使っていくかなど。
- データ解析に至るまでに必要な、被験者のリクルート方法、撮像方法、放射線技師との連携、データの前処理など教えていただきたいです。
- トラクトグラフィー
- トラクトグラフィーでお願いします。
- トラクトグラフィーの実際.特にdTVやDTI(DSI)studio,,TrackVis,MRTrix3等,トラクトグラフィーのソフトが乱立しているのでどれを使用したら良いのかわからない.
- トラクトグラフィーや深層学習のチュートリアルがあると嬉しいです。
- トラクトグラフィについて、基礎的な部分から研究への応用まで、取り上げていただければ幸いです。
- トラクトのグループ解析はどうしたらいいのか、手探りでやっているので、他の先生方がどうしているのか興味があります。
- トラクトはいろいろと信頼性の問題もあるのでしょうが、ぜひ、また勉強したいです。
- トラクト解析を勉強したいです。
- プランには記載されていたのですが、クラスター解析についてもぜひ受講したいと思っていました。
- ミエリンマップの解析。
- みなさんの実際の解析環境(ハードウェア等)について知りたいです。仮想環境、リモートデスクトップ等、工夫されていることがありましたら、参考にさせて頂きたいです。
- 画像解析の結果表示で注意すべき点などもう少し教えていただきたい。
- 画像研究の論文の批判的な読み方(例えば、査読者はどういう点に注意を払うか、など)について学べる機会があると嬉しいと思いました。
- 画像統計解析についてもう少し時間を取っていただくと良いと思います。
- 皆さんオススメの解析パイプラインなどあればそちらの概要が知れると嬉しいと思いました.
- 核医学画像の解析
- 簡単で結構ですので,PCのOSについての解説があると助かります.
- 器質性疾患において、右もしくは左の脳梗塞など局在に左右差がある場合の工夫、解析方法など知りたいです。
- 機械学習と画像解析について。
- 機械学習について、とりあげていただけると嬉しいです。
- 機械学習も何度かあったとおもいますが、なかなか理解が難しいところなので演習があればうれしいです
- 客観的な解析方法に思えるMRI解析でも解析者の主観的な判断(仮説や既報告による)が必要になるステップや、経験の多い先生が判断されるときの常識的な話とかも盛り込んでいただけると興味深いです。
- 具体的な発想はありません。
- 結果が出た後の、可視化の方法。
- 現在CONNでのResting-state fMRI解析に難渋しておりまして、取り扱っていただけると幸いです。
- 公共DBのデータの扱い方
- 昨年度のチュートリアルでCONNを取り上げて頂きましたが,マシンの性能の問題で途中で解析についていけなくなりましたので,今回のようなやり方で再度取り上げて欲しいと思いました.
- 撮像方法です。論文に載っている方法(マルチバンドなど)が、臨床機でできないことがあります。どうすれば?そんな疑問に答えます!という内容です。データ解析がいくら素晴らしくてもやはり撮像が最も重要だからです。
- 子どもの脳画像解析についてお願いします
- 資料の作成等、かなり労力のいるものと想像します。普段、講師やチューターの先生方が画像解析情報の更新をどのようにしているのか知りたいです。
- 取り上げられていましたが,統計解析の講義で,多重性とクラスター解析についてはもう少み詳しく説明を伺いたかったです.
- 取得した画像やアトラス等のデータを自在に加工する方法
- 縦断や反復測定の撮像を行う際,解析で個人間の比較とは異なる方法が必要であれば,取り上げていただきたいです。
- 縦断研究の取り扱いの詳細、実際にROIの値などを使用して統計解析する際(Rやmatlab)での処理の方法など
- 縦断的解析
- 小児の解析、infantFSなどについても紹介していただける回があるとありがたいです。
- 新しい技術に関する情報などございましたら、また教えていただきたく存じます。
- 川口先生のご講義の後半部分(FWE/FDRやクラスターレベル検定)について、もう少し詳しくお聞きできればと思います。
- 川口先生の後半の講義をじっくりお聞きしたいです。
- 川口先生の資料の後半の内容について詳しくお話を伺いたかったです。
- 川口先生の脳画像データにおける統計解析の講義をもっとガッツリと受けてみたいです.
- 全脳解析で有意な箇所がなかった場合、全く結果が報告できなくなってしまうのか、そこらへんがわかりませんでした。惜しくて有意じゃないのか、全然有意じゃないのか、そういう現場的な部分のコツやテクニックもご教示頂ける機会があれば、なお良いと思いました。
- 多施設共同研究やデータ共有の際にT1画像から顔を復元できないように画像の処理を行うと思います。そのやり方など、教えてもらえると嬉しいです。
- 大量のデータがある時に、例えばDTIの事前解析でコードを一つ一つ手作業で行うよりも別の方法はありますか。
- 知らないことだらけなので、その年のupdateを聞いていけるだけでたいへんありがたいです。
- 当研究施設では最近ミエリンマップの撮像も始めました(GEのqMRI、MAGiCシーケンスを取り入れています)。MAGiCは特段、前処理など必要もないのですが、ミエリンマップの概論や集団解析など、もし機会があれば教えていただきたいです。
- 頭蓋内器質性疾患があることの解析方法が学べてたいへん勉強になりました。
- 匿名化の部分で顔表面の情報を消す方法(国際脳のDBなどで検討されているもの)なども含めて教えて頂けると幸いです.
- 入門としては良いのではないかと思いました。
- 脳の容積と活動の大きさの関係
- 脳画像解析ソフト間の変換について、もう少し具体例を交えて、ゆっくりご講義をお聞きしたいです。
- 脳画像解析のソフトウェアが多く,使い分けがわからなくなることがあるため,それぞれのソフトウェアをどういった位置づけで使っているのか,使い分けをしているのかを知れる機会があれば嬉しいと思いました.
- 脳画像解析の統計のところで、どの場合はどの値を報告する、などの細かいところをご教授いただけますと幸いです。
- 脳幹の細部を選択する独自のテンプレートを作る方法を知りたかった。
- 脳機能画像の深層学習、機械学習などへの応用について
- 脳形態画像、拡散画像以外の脳画像解析についても取り上げていただくとありがたいです。
- 病巣と症状の関連を見る最新の解析方法。
- 本日の内容につきましては目標を考えると大変十分に思いました。復習をしっかりしたいと思います。
- 林先生のご挨拶の中であった,T1/T2の出し方などが知りたいです.
- 林先生のご挨拶の中であったような比較的新しい解析技術の話も聞いてみたいです.
- 臨床的に撮影された拡散強調画像でも実施可能な解析。
- 論文に使える画像表示の仕方をもっとききたい。
- 論文作成にあたり注意すべき事項などがあれば教えていただきたいです。
- 論文等で使用されている,●●人分のMRIデータを合成した画像(表現が適切でないかもしれません,申し訳ありません.)など,実際に論文を書く際に使われている解析方法についてさらに勉強したく思います.
- 齧歯類等の非臨床で実施する際のアトラス等の変更手順
- 1~3年経験の初級者でも理解できるようなMRIやfMRIの基本原理的なレクチャーも扱って頂けると嬉しいです.
- 1コマで良いので,解析PC(ハードウェア,仮想化)についての講義があると助かります.
- 3DT1画像のQCに関して,先生方がどのように普段確認されているのか気になりました.
- ABiSの内容から離れてしまうかもしれませんが、EEG解析などはやってもらえないでしょうか?"
- ANTs,SPM
- ANTsの使い方を教わりたい!
- B0マップを解析に取り込む方法です。B0マップの扱い方を、ぜひハンズオンで教えていただけたらありがたく存じます。
- BIDSについてより詳しく知りたく思います。
- CAT12についての最新のチュートリアルがあるとうれしいです(縦断解析含め)。
- CAT12の使い方に加え、MEGやSPECT、PETの解析など少しでも扱っていただけるとうれしいです
- CONNの使い方など
- CONNを使用した脳画像解析方法、子どもの脳画像解析のポイント
- CONNを用いたFCの解析について、解析本にない内容について、取り上げてほしいです
- Deep learning、Machine learningを使用した画像解析
- DSI studioなど、GUIでのtractographyやROI解析など
- DTIとミエリンマップの比較にも興味があります。
- DTIによるstructural connectivityやtractgraphyをご教授いただけましたら幸いです。ネットワーク解析をしている論文を見かけるのですが、初学者にはハードルが高い印象です。
- DTIのTBSSなどの縦断解析についてぜひ知りたいです。よろしくお願いします。
- DTIのtract-based analysis(FreeSurfer_TRACURAなど)
- DTIのトラクトグラフィーについて、もう一度学びたいです(去年Slicerの講義があったのですが)。
- DTIの講義ではb0を位相エンコーディングを変えて何度か撮像したほうがよい、など撮像方法へのアドバイスもありましたが、解析ではなくデータ取得に先立って考えなければならない点、などの内容も今後検討いただけたら非常に助けになりそうです。
- DWIの解析でTBSS以外の解析方法があれば取り上げて欲しい
- DWIやfunctionalが多いようですが、それ以外の画像解析もできればお願いします
- FA画像やNODDIについてとりあげてほしいです
- Fieldmapについて詳しく知りたいです。
- fMRIのネットワーク解析
- fMRIの解析について学びたかったので、来月のチュートリアルをとても楽しみにしております!
- FreeSurferではQCのより実際的な良い例と悪い例などを比較し、どの程度までを許容して解析を進めるか等の一指針のようなものがあると大変助かります。
- FreeSurferとBrainSuitesとMRICloudとの比較が知りたいです
- FreeSurferとBrainSuiteの比較が知りたいです
- FreeSurferとFSLを使った研究のfuture direction、最新状況について
- FreeSurferの縦断解析、CAT12のVBM
- FreeSurferの縦断解析やTRACULA、FS-FASTを伺いたいです
- FSLを用いた画像の補正について。
- function MRI
- function MRIの処理
- gitの利用方法
- GLMの交互作用
- GPUを使う場合の処理について
- HCP workbenchについて環境構築や使用方法についてレクチャを希望します。ミエリンマップの画像作成方法について知りたいです。
- HCPのマルチモーダルな画像解析、Workbench、ミエリンマックの作成方法が知りたいです。
- HCPや国際脳データベースの前処理やパイプラインを使った解析に関して興味があります。
- Human Connectome Projectの解析です。HCPのデータを使って、HCPのpipelineで解析するという内容です。
- ICA解析について基礎から学びたいです。
- JSONファイルから必要な情報を引き出す効率的な方法があったらうれしいです。
- lead DBSやTHOMAS, VentricleWMHsegmentationなどgithubやdocker環境で提供される画像解析ソフトの使用法を解説していただけたら幸いです。
- Lin4Neuroの海外、国際的な認知度や利用状況、今後の展望など、Lin4Neuroについて詳しく知りたい
- MRI(VBMやtask fMRI)以外の解析
- MVPAなどの一部を取り上げていただけるとありがたいです
- NODDI、GBSSについて可能であれば、ご教授頂きたいです
- NODDIの解析について
- NODDIの解析について講義していただけると大変嬉しいです。
- Pythonでの解析が進歩してきているので、それらを使った解析。Nipypeなど。
- Pythonに特化したスクリプティングの講義も、ぜひよろしくお願いします。
- Pythonの基礎についてもチュートリアルがあると嬉しく思います。
- recon-allの結果のQCを複数の実例を用いてじっくり行い、修正前後の画像を比較検討するなどの機会があったらいいなと思いました。受講者には事前に20例ほどのデータが配布されて、各自事前宿題としてrecon-allとQCをやっておく→講義当日はそれぞれの画像のどこが問題だったか、どう直すか、recon-allやり直し後はどう改善されていたか、を1例ずつ解説していく…などが理想です!
- resting state fMRIの解析方法について
- Resting stateにおける2群間の縦断データ比較のように、複数の水準、要因を比較する際の分析の仕方
- rsfMRIの解析
- RとPythonの応用など
- RやPythonの使い方
- SPECTではなく、CT perfusion dataを用いてFSLやSPMで解析することは可能なのでしょうか。脳循環画像としてCT perfusionを用いることが多く、有用性がありそうだと感じているのですが、解析できれば大変うれしいです。
- SPECTや脳波、MEGなどのモダリティの解析法が知りたいです
- SPM12(MATLAB)で活用できるスクリプトなど
- SPM12、FSLを用いた前処理の比較について。
- SPMでしょうか?
- SPMの使い方
- SPMやCATについての講義をしてほしいです。根本先生のSPMの本を読みながらさせていただいていますが、チュートリアルで取り上げて頂けると普段気になっていることが聞けるのでありがたいです。
- SPSSの使い方、SPSSでの統計解析の仕方、代表的な画像解析/AIソフトのおおまかな解説
- SynthSRの紹介がありましたが、とても興味深く思いました
- TBSSでの相関関係の表示方法
- TBSSの追加解析のスクリプトの詳細について、もう少しゆっくり話を聞きたかったです。
- TBSS以外のDTIのROI解析やTractgtaphy
- Tractography
- tractographyの実践について
- traculaについて知りたいです。
- VBM
- VBM、根本先生の教科書には載っていない縦断解析について
- VBMやFCなどで、結果の表示のfigureなどの最近のやり方はありますか?
- VBM解析、rs−fMRI解析についてお願いできると幸いです。
- VLSM
- vlsm解析
- Workbench
- XTRACT
- XTRACTによるトラクトグラフィー XTRACT_STATS , XTRACTblueprintの有用な使用方法について
その他、ご意見ありましたら自由にご記入下さい。(抜粋)
- アナウンスをなさる際,マルチモニタなど,受講環境もアナウンスなさると良いと思います.
- アンケートの回答が遅くなり、大変申し訳ございませんでした。今週末もたいへん楽しみにしております。
- いつも、本当に有難うございます。自学自習はできないところが多い脳画像解析につきまして、「どうして、そのようにするのか」の部分を含めて、ご講義いただけること、大変ありがたいです。
- いつもありがとうございます。
- いつもいつもありがとうございます.講義をされた方々は3日間の講義期間だけではなく,それに至るまでの準備期間も相当な労力をかけておられているとお察しいたします.
- いつもお世話になっております.本当にありがとうございます.
- いつもご丁寧なご教授をいただき,大変感謝しております.
- いつも大変勉強になっています 講師の皆様に心から感謝申し上げます
- いつも丁寧な準備とご講義をありがとうございます。次回以降も是非参加させていただけたら幸いです。ありがとうございました。
- いつも本当にありがとうございます。ABiSの支援が継続することになったのは本当に嬉しいです。
- オンラインでこれほど素晴らしいチュートリアルはございません。講師の先生方の事前のご準備の賜物です。今後も続くとのこと、素晴らしいです。本当にありがとうございました。
- オンライン開催は、遠方在住の私にとってありがたいことしかなく、大変感謝しております。
- お忙しいところ,本当に素晴らしい講義をありがとうございました.実になるように,しっかりと事後学習もさせて頂ければと思います.
- ここまで手厚いサポートをしていただけるチュートリアルは知らないかったので、解析だけでなく、運営に関しても大変勉強になりました。
- ここまで丁寧にご説明していただいているチュートリアルは初めてでしたので、非常に感激致しました。先生方のご準備も非常に大変だったと推察されます。
- この3日間、お疲れ様でした。内容が難しくても先生方の細かい説明や対応のおかげで諦めずに最後まで参加できているので、今後ともどうぞよろしくお願いします。
- このような機会をもうけていただきまして、講師やチューターの先生方に大変感謝しております。
- このような講習会が年に2度くらい開催されるといいなと思います
- このような充実したチュートリアルを無料で受けさせていただけることに驚いております。
- このような勉強会(ご講義)はとてもありがたいです!
- この度はとても貴重な機会をいただきありがとうございました。今後ともよろしくお願い致します。
- コピー機能が便利すぎて自分ができるようになったと勘違いしそうでしたが、ターミナルに入力することで自分の実力がよく分かり、これからしっかり勉強しようと思いました。非常に有意義な時間をありがとうございました。
- コマンドラインのPC操作がMS-DOS時代からなので懐かしい感じがした
- これからもぜひ、日本のneuroimagingの底上げになるよう、宜しくお願い致します。
- ご講義くださった先生方、チューターの皆様、本当にありがとうございました!
- スタッフの方々も本当に大変だと思います.皆さまのご恩に報いるためにも学んだことを自分の研究にしっかりと活かして参りたいと思います.
- ターミナルにタイプするのが追い付かなかったり,ミスタイプがあったため,追い付くことに必死になりました。動画を拝見し,しっかり復習したいと思います。
- チュートリアルの資料や準備お疲れ様です。研究法を取得するとても貴重な講習会であると思います。今後もよろしくお願いいたします。
- チュートリアルの実施,誠にありがとうございました。
- チュートリアルの準備をする段階で、何回か問い合わせをして、その都度、根本先生に回答頂き、大変恐縮でした。お一人で様々な質問に対応されているのかと思い、本当に頭が上がらない思いでした。
- データ解析結果をもとに論文を書く段になって、細かな解析の追加必要性に気づく(しかし方法がわからない)ことも多く、直接質問でき即時にご意見をいただけるこの機会は、本当にありがたく、ぜひ継続してほしいと思っています。
- どうもありがとうございました。先生方、チューターの皆様には深く感謝申し上げます。貴重なご示唆をくださった参加者の皆様にも御礼申し上げます。
- とても濃密な内容で講習を開催していただけたこと、講習に参加させていただけたことに心より感謝いたします。
- とても有意義な講義で、絶対に自分では理解できないところまでわかりやすく解説していただき本当に有難いです。感謝しかありません。
- とても良いです! どうもありがとうございました
- とにかく、先生方に感謝いたします。準備も大変だったことと思います。
- ワークショップが終わった後も、仲間内で自由に質問や情報共有ができる場所があると嬉しいです。
- わかりやすい資料と丁寧な説明をありがとうございました。感謝申し上げます。
- 一部病院に呼ばれて参加できませんでしたが、非常に充実した内容で参加してよかったです。よく復習したいと思います。どうもありがとうございました。
- 下地先生のセッションは、芸術の領域と感じました。
- 何度か解析しているVBMのお話は非常に深く理解できました。一方で解析をしたことのない分野は講義ではついていくのが難しかったです。
- 解析のチュートリアルで有益な講義をありがとうございます。大変助かっています。今後ともよろしくお願いいたします。
- 皆様の御尽力に感激しております。素晴らしい講座をありがとうございます。
- 拡散ならTRACULA、restingならFSLやDPABIでの処理やsurface-basedの解析、taskならMVPAなどなど、これまで行われていない内容のチュートリアルがあったらいいなと思います。
- 拡散画像の講義は、新しい内容が多くてためになりました。
- 感謝しかないです!!! ありがとうございました
- 関連する勉強会や学会なども参加してみたいなと思いました。そのようなものがあればご教示いただければ嬉しいです。本日は本当にありがとうございました!
- 丸1日の講義ありがとうございます!非常に勉強になる貴重な1日でした.
- 帰国後すぐの参加で疲労困憊でしたが、無理して参加した甲斐がありました。ありがとうございます。
- 貴重なお時間有難うございました。
- 貴重なご講義ありがとうございました。
- 貴重なご講義をありがとうございました.
- 貴重なご講義をありがとうございました.講師の先生方,チューターの先生方に,大変感謝しております.
- 貴重な機会、ありがとうございました。根本先生はじめ皆様のご厚意に、感謝します。
- 貴重な機会ありがとうございました。
- 貴重な講義をありがとうございました。大変勉強になりました。
- 研究の幅が広がったように感じます.
- 講義された先生方、チューターの先生方本当にありがとうございました。
- 講師、チューターの先生方、3日間誠にありがとうございました。
- 講師の先生方におかれましては、いつも素晴らしい講義を頂き本当にありがとうございます。
- 講師の先生方には本当に感謝申し上げます.
- 今回、2日目のPMがどうしても外せない仕事のため受講できず大変悔しい思いでした。必ずビデオで受講させて頂き、機会を活かしたいと思います。
- 今後とも,ご指導ご鞭撻のほどよろしくお願いいたします.
- 今後とも何卒よろしくお願いいたします.
- 今後の研究につなげられるように,復習に努めたいと思います.
- 今後の仕事に役立てればと思います。
- 今後も,このような機会がありましたら,参加させていただきたく存じます.
- 今後も勉強させて頂きたいです。よろしくお願いします。
- 今後も末永く継続していただけることを切に願っております
- 今年で3年目の受講となりました、毎回、講義内容やシステムがパワーアップしており、素晴らしいなと感謝しております。講師、チューターの先生方、ありがとうございました
- 根本先生、本当にありがとうございます。
- 参加した時は大変勉強になりましたのでまた参加させていただきたいです。
- 参加するたびに新たな発見があります。海外で開催されるワークショップにもたくさん参加しましたが、いつもABiSが一番わかりやすいと思います。しかも無料なので、今後もオンラインでたくさんの参加者を受けいれていただければと思います。
- 仕事の都合であまり参加できなかったため、動画配信で学習させていただきます。
- 仕事の都合で参加が難しく、後日動画を共有していただけるとこのとでしたので、大変ありがたいです。
- 死後脳の画像解析を実施された先生はいらっしゃいますか。
- 私自身もいつか少しでも、どなたかに解析方法をお伝えできるくらいに、知識を身につけねばと思います。精進します。
- 事前準備からわかりやすく対応していただきまして、感謝しております。
- 事前準備から当日のチュートリアルまで相当なタスクだと存じますが,無料でオンラインで公開していただいて大変ありがたいです.
- 事前準備のご苦労は相当なものだと思いますが、このような貴重な機会をいただけたことに感謝しかありません
- 次回もよろしくお願い申し上げます。
- 実際に打ち込むことで勉強になる部分も多く,コマンドにもっと慣れておく必要があったと思いました。
- 受講者は少なるかもしれませんが、動物用MRI画像の解析セミナーがもしあればぜひ受講したい!
- 受講料払ってでも参加したいチュートリアルだと毎回感じています。
- 十分な時間をかけて基本的な操作などを学ぶことができ、大変勉強になりました。ありがとうございました。自分の研究にどのように活かしていくか、これから検討していきたいと思います。
- 準備にも講義にも大変な労力をかけてくださり、本当にありがとうございます。
- 初回からほぼ毎回参加させていただいています。内容はもちろん、開催方法などどんどん進化していくのでとても勉強になります。これからも継続されることを願っています。
- 初学者用と銘打たれた今回の講習会でしたが、何度か参加させていただいている自分でも、新鮮な学びが多くある回だったというのが率直な感想です。
- 初心者でも理解しやすい内容でした。ありがとうございました。
- 将来、自分たちでチュートリアルを作ろうとした時は、今回のチュートリアルを参考にしたいと思います。誠にありがとうございました。
- 上田先生の資料が年々分かりやすさを増していると感じました。本当に分かりやすかったです。
- 新型コロナに罹患しておりまして、Webセミナーだからこそ参加できました。
- 誠にありがとうございました。
- 先生の操作を見ながら、自分のPCの操作をする際に、自分のPCがうまく動かなかったり、処理が遅いと途端に迷子になって、おいてけぼりになってしまうのが困りました。
- 先生方,チューターの皆様に感謝申し上げます.
- 先生方、3日間、本当に有難うございました。大変勉強になり、受講させて頂けましたことに感謝しております。
- 先生方、本当にありがとうございました。このチュートリアルは長く続けていただけましたら幸いに存じます。
- 素晴らしい講義、テキスト、チュートリアルありがとうございました!
- 多くのデータや仮想空間を無料で提供していただき,本当にありがとうございます.
- 多少、難しい内容がありましたが、このような高水準の講義を続けられることが、確実に日本の研究レベルの底上げとして結実するのだと期待します。
- 体調不良等で参加できなかったところのもあるのですが、そのビデオを視聴しつつ、次回のチュートリアル楽しみに待ちたいと思います!
- 大変お疲れさまでした。
- 大変貴重な機会をいただけていること心より感謝しております。
- 大変参考になりました。ありがとうございました。
- 大変濃密な内容のチュートリアルで大変満足致しました。お忙しいところ3日間、講師の先生方、チューターの先生方に感謝申し上げます。誠にありがとうございました。
- 大変勉強になりました、有り難う御座いました。
- 大変勉強になりました。ありがとうございました。
- 大変勉強になりました。どうもありがとうございました。
- 大変勉強になりました。今後ともよろしくお願い致します。
- 知った気になって作業に当たっていたのかもしれないという気づきも多々あり、自分のしている解析方法をきちんと理解する姿勢を持ち続けることは本当に大事だと感じました。
- 丁寧なチュートリアルを無料で受講できて大変有用だった
- 丁寧に説明して下さりよく理解することが出来ました。本当にありがとうございました。
- 長い時間,講師の先生や主催されている先生方は非常に大変かと思います.しっかり勉強したことを,研究に還元したいと思います.
- 長時間にわたり,建設的にサポートしていただき,深く感謝申し上げます。
- 長時間に渡り、大変充実した内容の講義をありがとうございました。
- 土日子どもが熱を出し,ほとんど参加できず,アンケートもほとんど意味のないものになって申し訳ありません。録画を共有いただいた後,しっかりと確認したいと思います。
- 匿名化の仕方、統計、コマンド入力の基本など重要だけど教わることができない点をしっかりとりあげていてよかった。
- 特にありません。
- 特になし
- 難しくついていけない時もありましたが、また動画を見返してしっかり復習したいとおもいます
- 日曜日の長時間に渡りこのような場を設けていただき、大変だったかと存じます。私は上司から偶々教えてもらったのですが、参加できて本当に幸運でした。次回も絶対参加します。
- 脳画像解析に対して漠然としたハードルを感じ、自分には無理かなとしか思っていませんでした。今も難しいなと思っていることは変わりありませんが、これを機会に挑戦しようかなと前向きになっています。
- 脳画像解析自体にほとんど接したことがなかったので、単語レベルで分からない/知らないことが多く講義についていくのが精いっぱいでしたが、たくさんの刺激を頂き新しい世界を垣間見ることができました。
- 非常に充実した内容でした。どうもありがとうございました。
- 非常に濃い内容で3日間非常に充実した時間でした。
- 非常に濃い内容で大変勉強になりました。脳画像解析に関して難しく躊躇していたところがありましたが,大変理解が深まりました。
- 非常に有益な講習会で助かりました。また次回も是非参加させていただきたいと思います。今後ともどうぞ宜しくお願い致します。
- 普段学ぶことができないことを、基本的なところからお話いただけたので、大変勉強になりました。本当にご準備ありがとうございました。
- 本当にありがとうございます。このチュートリアルで学んだ事を、研究で発揮していきます。
- 本当にお疲れ様です
- 本日はお忙し所、大変手厚いチュートリアルを誠にありがとうございました。次回も大変楽しみにしております。よろしくお願いいたします。
- 毎回充実した内容で運営スタッフの皆様には大変感謝いたします。今後も是非継続されますようよろしくお願いいいたします。
- 毎年楽しみにしております、今後とも宜しくお願い致します。
- 毎年年末年始のこの講義を楽しみにしています。先生方、準備等大変だと思いますが、どうかぜひ、続けてください。
- 12/11の講義のおかげで2日間の講義の理解がしやすかったです。ありがとございました。
- 1月もよろしくお願い致します。
- 3日間、本当にありがとうございました。講師の先生方に感謝してもしきれません。次の機会もぜひ参加したいと思います。
- 3日間ありがとうございました。
- 3日間どうもありがとうございました。復習して今後にいかしたいと思います。
- 4回目の受講になりますが、毎回変化とブラッシュアップを感じます。チューターの先生方の丁寧な対応に脱帽です。
- ABiSチュートリアルの受講受付のメーリスが届くたびに、今回もチュートリアル当日は、外は雪かなぁと思いつつ、楽しみにしています。
- BIDSはほとんど理解できていませんが、ぜひまた聞きたいです。ありがとうございました。
- fMRIの回では、gPPIの解析方法、縦断解析を知りたいです。臨床現場での研究ではニーズがあるように思います。よろしくお願いします。
- FreeSurferのコマンドなどあることすら知らなかったです。
- FreeSurferの集団解析に取り組んでいた所でしたので,今回の講義で学ことができ,大変参考になりました.
- Macネイティブでの環境セットアップで根本先生にサポートいただきました。ご多忙のところ本当にありがとうございました。
- MRIで撮像した画像をMRIから取り出す手段で効率的な方法などあったらご教示いただけると嬉しいです。
- MRIの原理について徹底的に理解する機会、fMRIprepなどのパイプラインで何が行われているかを細かく学ぶ機会も欲しいです。
- MRIの原理的な部分の理解が十分でなかったため、そこのところの知識を学びなおし、再度チュートリアルを見返したいと思いました。ありがとうございました。
2022年12月ABiS脳画像解析チュートリアル
【日 時】2022年12月11日(日)、17日(土)~18日(日)の3日間【会 場】完全オンライン
【参 加】事前登録制(参加費無料、PC環境が準備できない場合は受講できないことがあります)
【概 要】
12月11日(日) 脳画像解析入門
08:30 Zoomオープン
09:00-09:20 開会、オリエンテーション
09:20-10:10 第1部(1) 画像ビューワー (筑波大・根本)
10:20-11:10 第1部(2) DICOM画像の理解 (根本)
11:20-12:10 第1部(3) DICOM画像のNIfTI画像への変換 (根本)
12:10-13:00 昼休み
13:00-14:00 第1部(4) 一般線形モデル (根本)
14:10-15:10 第1部(5) 脳画像統計 (佐賀大・川口)
15:20-16:10 第1部(6) シェルスクリプト (根本)
16:25-17:45 第1部(7) BIDS (根本)
17:45-18:00 質疑応答
12月17日(土) FreeSurferチュートリアル
08:30 Zoomオープン
09:00-09:20 開会、オリエンテーション
09:20-10:10 第2部(1) FreeSurferの概要 / recon-all (筑波大・根本)
10:20-11:10 第2部(2) Freeview / ROI解析 (根本)
11:20-12:20 第2部(3) グループ解析 (根本)
12:20-13:10 昼休み
13:10-14:10 第2部(4) Slicerを用いたQC (岩手医大・山下)
14:20-15:20 第2部(5) Slicerを用いたトラブルシューティング (山下)
15:30-16:30 第2部(6) Freeviewを用いたトラブルシューティング (山下)
16:40-17:40 第2部(7) 他のソフトとの連携 (根本)
17:40-18:00 質疑応答
12月18日(日) 拡散MRIチュートリアル
08:30 Zoomオープン
09:00-09:10 開会、オリエンテーション
09:10-10:10 第3部(1) DWIの前処理 (慶応大学病院・上田)
10:20-11:20 第3部(2) TBSS (Non-FA Analysisも含む) (上田)
11:30-12:30 第3部(3) 脳器質的疾患のTBSS (上田)
12:30-13:20 昼休み
13:20-17:00 第4部: TBSS解析後の結果表示と追加解析
13:30-14:30 第4部(1) FSLeyesのアトラスツール (順天堂大・下地)
14:40-15:40 第4部(2) bashの活用 (下地)
15:50-16:20 第4部(3) all_FA_skeletonisedの活用 (下地)
16:30-17:00 第4部(4) 関心領域の作成 (下地)
開催済みの脳画像解析チュートリアル
2022年1月29日~30日 第11回ABiS脳画像解析チュートリアルを開催致しました。
2021年12月18日~19日 第10回ABiS脳画像解析チュートリアルを開催致しました。
2021年1月30日~31日 第9回ABiS脳画像解析チュートリアルを開催致しました。
2020年12月19日~20日 第8回ABiS脳画像解析チュートリアルを開催致しました。
2020年2月8日~9日 第7回ABiS脳画像解析チュートリアルを開催致しました。
2019年3月2日~3日 第6回ABiS脳画像解析チュートリアルを開催致しました。
2018年12月22日~23日 第5回ABiS脳画像解析チュートリアルを開催致しました。
2018年3月3日~4日 第4回ABiS脳画像解析チュートリアルを開催致しました。
2018年1月20日~21日 第3回ABiS脳画像解析チュートリアルを開催致しました。