ゲノムワイド関連解析による膵がんの新規感受性遺伝子GP2の同定
愛知医科大学医学部公衆衛生学
林 櫻松
Lin Y, Nakatochi M, Hosono Y, Ito H, Kamatani Y, Inoko A, Sakamoto H, Kinoshita F, Kobayashi Y, Ishii H, Ozaka M, Sasaki T, Matsuyama M, Sasahira N, Morimoto M, Kobayashi S, Fukushima T, Ueno M, Ohkawa S, Egawa N, Kuruma S, Mori M, Nakao H, Adachi Y, Okuda M, Osaki T, Kamiya S, Wang C, Hara K, Shimizu Y, Miyamoto T, Hayashi Y, Ebi H, Kohmoto T, Imoto I, Kasugai Y, Murakami Y, Akiyama M, Ishigaki K, Matsuda K, Hirata M, Shimada K, Okusaka T, Kawaguchi T, Takahashi M, Watanabe Y, Kuriki K, Kadota A, Okada R, Mikami H, Takezaki T, Suzuki S, Yamaji T, Iwasaki M, Sawada N, Goto A, Kinoshita K, Fuse N, Katsuoka F, Shimizu A, Nishizuka SS, Tanno K, Suzuki K, Okada Y, Horikoshi M, Yamauchi T, Kadowaki T, Yu H, Zhong J, Amundadottir LT, Doki Y, Ishii H, Eguchi H, Bogumil D, Haiman CA, Le Marchand L, Mori M, Risch H, Setiawan VW, Tsugane S, Wakai K, Yoshida T, Matsuda F, Kubo M, Kikuchi S, Matsuo K.
Genome-wide association meta-analysis identifies GP2 gene risk variants for pancreatic cancer
Nat Commun. 2020 Jun 24;11(1):3175. doi: 10.1038/s41467-020-16711-w.
https://www.nature.com/articles/s41467-020-16711-w
プレスリリース https://www.pref.aichi.jp/cancer-center/ri/news/topics.html
膵がんは、近年増加傾向にあるにもかかわらず、発生要因の大部分は不明です。 双生児の研究では膵がん発症原因のうち遺伝要因が約36%を占めることから、遺伝要因の果たす役割は大きいと考えられています。膵がんの発症に関連するコモンな遺伝的変異(主にSNP)の同定は、ゲノムワイド関連解析(GWAS)が有効とされています。これまで主に欧米人集団を対象としたGWASから、ABO血液型など膵がん感受性遺伝子領域計が23か所報告されてきました。しかしながら、日本人を対象としたGWASは1件報告されているものの、一般的なゲノムワイド有意水準(p<5×10-8)に到達したSNPは同定されていませんでした。そこで、本研究グループは、アジアで最大規模の膵がんGWASメタ解析(症例2,039人、対照32,592人)を実施しました。その結果、3カ所のゲノム領域(13q12.2, 13q22.1, 16p12.3)において、ゲノムワイド有意水準を満たす関連を認めました。特筆すべきは、16p12.3は新規膵がん感受性領域であるということです(図1)。
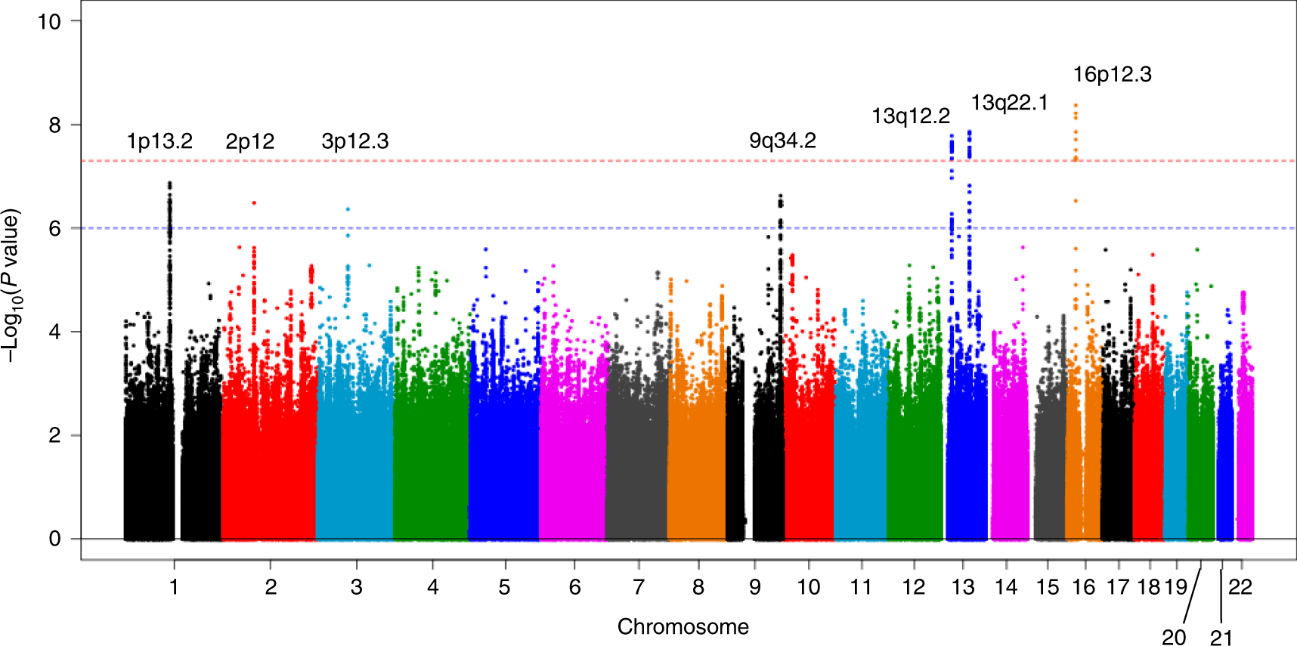
図1 マンハッタンプロット。横軸に染色体番号、縦軸にlog10(P値)を示す。
(http://creativecommons.org/licenses/by/4.0/ 以下同)
16p12.3にて膵がんリスクと有意な関連を示した10個のSNPは、全てGP2という遺伝子領域内に存在していました(図2)。そのうち、最も強い関連を示したSNP rs78193826は、GP2のコーディング領域(GP2蛋白質に翻訳される領域)に存在していました。このSNPのマイナーアレルTを持つ者の膵がんリスクは、持たない者と比べて約1.5倍に高まることが分かりました(オッズ比:1.46, 95%信頼区間:1.29-1.66)。さらにこのSNPは、CアレルからTアレルに変わることでアミノ酸変異(Val→Met)を伴い、GP2蛋白質の構造や機能に有害な影響を与えると予測されました。これらの結果から、rs78193826はGP2遺伝子領域内の膵がんの発症原因となる遺伝的変異(causal variant)である可能性が高いと考えられます。
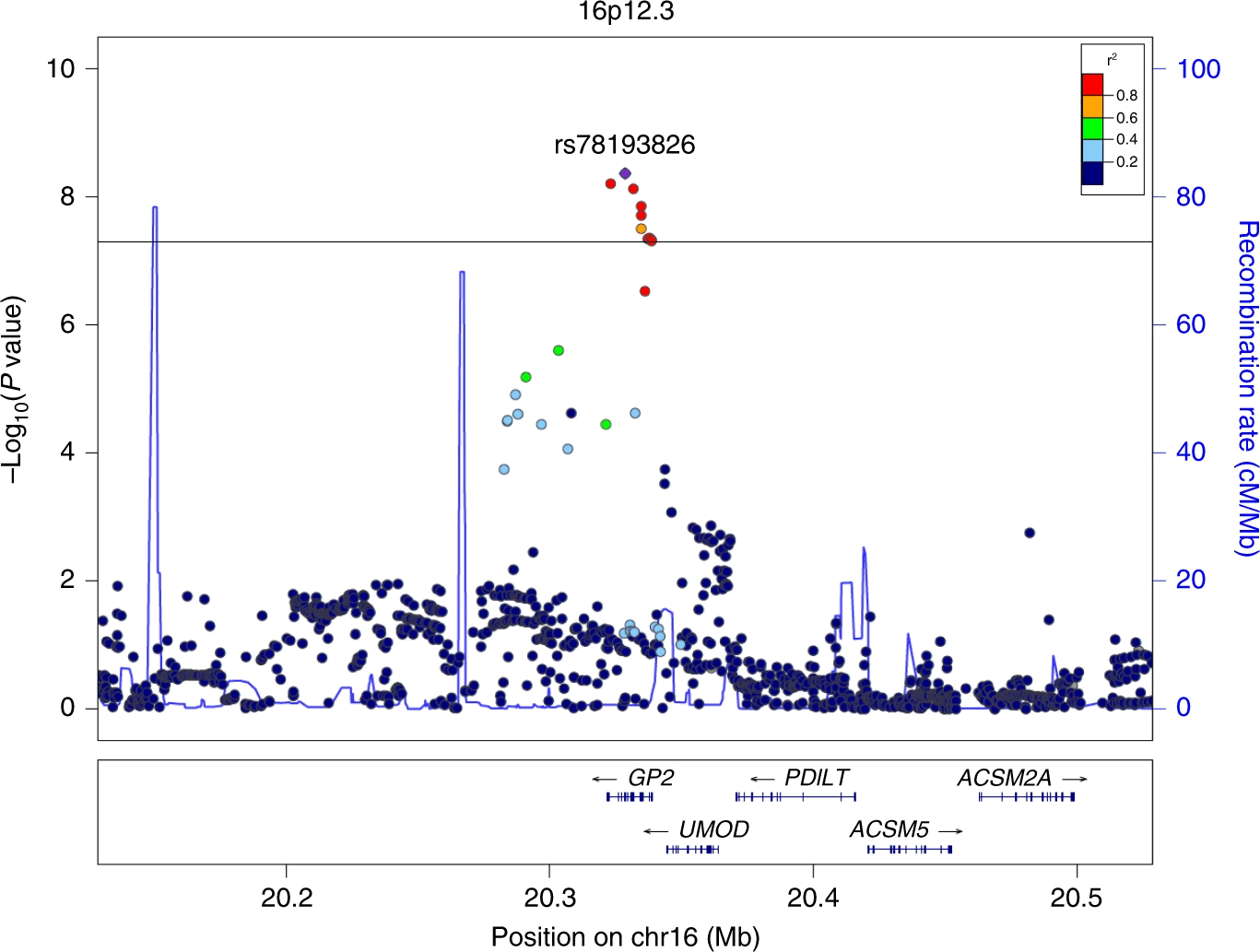
図2 膵がん感受性領域16p12.3の領域図(regional plot)。
横軸に16番染色体における位置、縦軸に-log10(P値)を示す。
GWASの成果を臨床へ応用するためには、統計学的関連だけでなく、causal variantに起因する疾患発症メカニズムの解明が欠かせません。本研究では、候補causal variantであるrs78193826に注目し、膵がん細胞株PATU8988S、ゲノム編集(CRISPR-Cas9)、次世代シークエンサー(RNA-seq)を併用した機能解析を行いました(図3)。まず、CRISPR-cas9でrs78193826の変異をPATU8988Sに導入し、GP2ゲノム編集細胞株(V282M株)を作成しました(図3a)。次に野生(WT)株とV282M株に対しRNA-seqを実施し、遺伝子セット解析(GSEA)により両株間の遺伝子発現プロファイルの違いを評価しました(図3b,3C)。その結果、数多くの有意な遺伝子セットが検出されました。特に、HALLMARK_KRAS_SIGNALING_DN等のKRASの活性化により発現が低下する遺伝子群が、GP2 V282株で有意に低発現していたことが確認できました。図3dは、HALLMARK_KRAS_SIGNALING_DN シグネチャーの遺伝子の発現を示すヒートマップで、KLK7、KLK8、BMPR1Bの3つの遺伝子が特にGP2 V282M株で低発現していました(図3d)。これら3つの遺伝子について、qRT-PCR で検証したところ、いずれも野生(WT)株と比べV282M株のほうが有意に低発現していました(図3e)。これらの機能解析の結果より、rs78193826はKRAS活性を調節することで膵がんリスクに影響を及ぼすことが示唆されました。
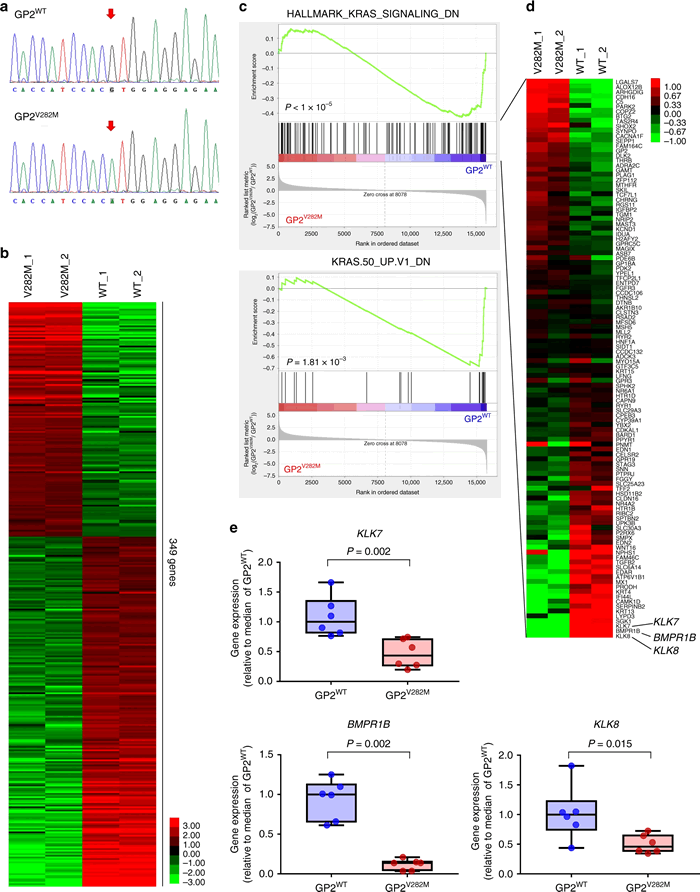
図3 GP2候補causal variant rs78193826の機能解析
今後、GP2領域の遺伝的変異と膵がん発症との因果関係を明らかにするために、fine-mapping によるGP2領域のcausal variantの確定や、動物モデルを用いたさらなる機能解析がとても重要であると考えます。GP2領域の遺伝的変異に起因する膵がん発症機序を明らかにすれば、膵がんの早期発見や創薬につながることが期待されます。
最後になりますが、本研究の遂行にあたって、コホート・バイオリソース支援(J-MICCによるreplication dataの提供)及び大規模バイオデータ解析支援(SNPデータのimputationやGWAS関連解析、RNA-seqデータ解析)をいただいたことに心より感謝申し上げます。