Research
- ウイルスによる腫瘍化メカニズムに関する研究
- がんと感染症のエピゲノムコードに関する研究
- がんや感染症のクローン進化に関する研究
- データサイエンスを駆使した新たな治療薬開発研究
- さまざまな感染症に関する研究 (東京大学UTOPIAとの共同研究)
- 主な研究費 (研究代表者のみ)
(1)ウイルスによる腫瘍化メカニズムに関する研究
ウイルス感染症の中には、長い潜伏期を経て重篤ながんを引き起こすものが存在します。私たちは、ヒトT細胞白血病ウイルス1型(Human T-cell leukemia virus type 1, HTLV-1)やエプスタイン・バーウイルス(EBV)を対象に、腫瘍化メカニズムの解明、および成人T細胞白血病リンパ腫(ATL)、HTLV-1関連脊髄症(HAM/TSP)、EBV関連疾患の研究を行っています。さらに、白血病・悪性リンパ腫などの他の血液腫瘍や、HIV-1感染における分子病態・潜伏化についても研究を進めています。
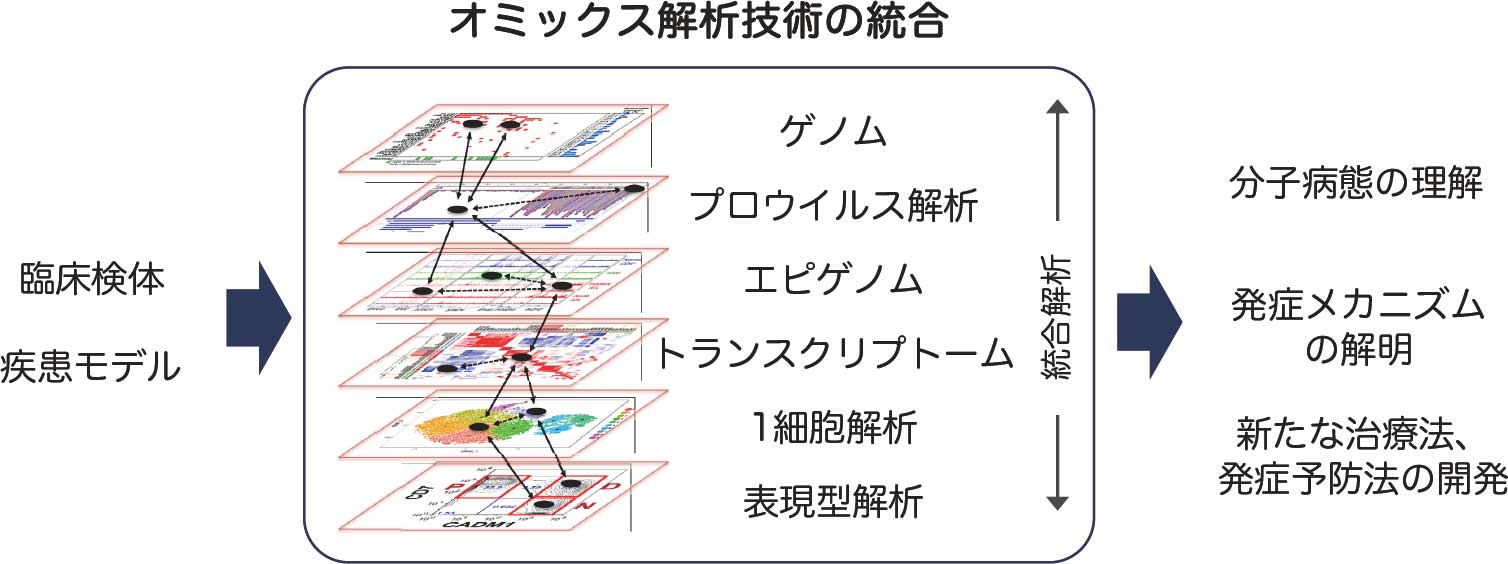
学内外の多くの研究者との共同研究によって、臨床検体を用いた次世代シーケンス(NGS)技術やシングルセル解析技術などを取り入れ、実験医学とデータサイエンスの両輪で推進することに特に力を入れています。臨床ビッグデータと統合オミクスを基盤に、発症機序の解明から早期診断・発症予防・新規治療の創出へとつなげる成果の発信を目指しています。
主な参考論文
- Yamagishi et al., Nat Commun. 12, 4821, 2021
- Fujikawa et al., Blood, 127, 1790-1802, 2016
- Yamagishi et al., Cancer Cell, 21, 121-135, 2012
(2) がんと感染症のエピゲノムコードに関する研究
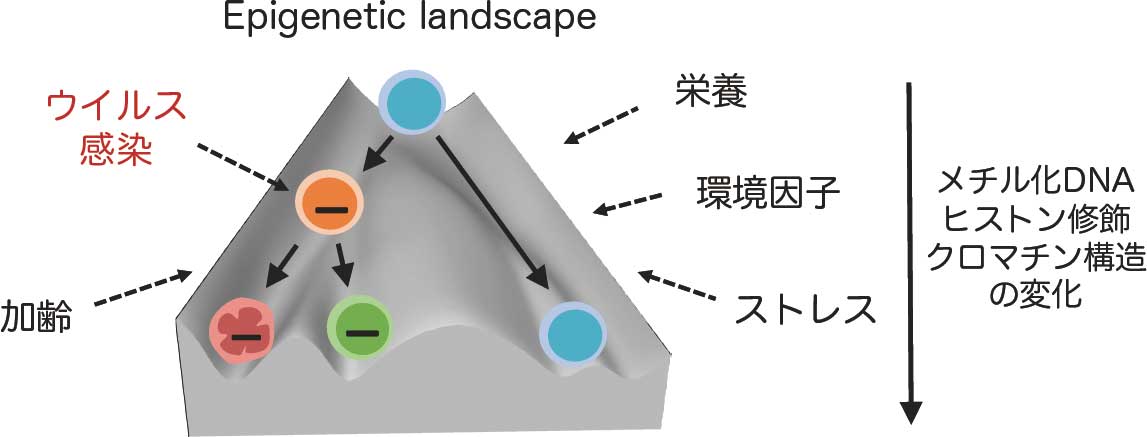
エピゲノムとは、遺伝情報(ゲノム)の使い方を規定する層であり、年齢や環境によって動的に変化します。私たちは、レトロウイルス感染が宿主細胞のエピゲノム(ヒストン修飾・DNAメチル化・クロマチン構造)を大規模に再配線することを明らかにしてきました。こうした変容は、感染によって宿主細胞に刻まれる本質的な改変であり、発症機序・分子病態・細胞運命制御に直結します。現在、がんや感染症で形成される異常エピゲノムの特徴・多様性・可塑性に注目し、ウイルス感染やがんに共通する「エピゲノムコード」の解読を目指して研究を進めています。
東京大学プレスリリース
- https://www.u-tokyo.ac.jp/focus/ja/articles/a_00458.html
- https://www.k.u-tokyo.ac.jp/information/category/press/8152.html
主な参考論文
- Yamagishi et al., Cell Rep., 29, 2321-2337, 2019
- Fujikawa et al., Blood, 127, 1790-1802, 2016
- Yamagishi et al., Cancer Cell, 21, 121-135, 2012
- Yamagishi et al., Nature, 627, 221-228, 2024
(3) がんや感染症のクローン進化に関する研究
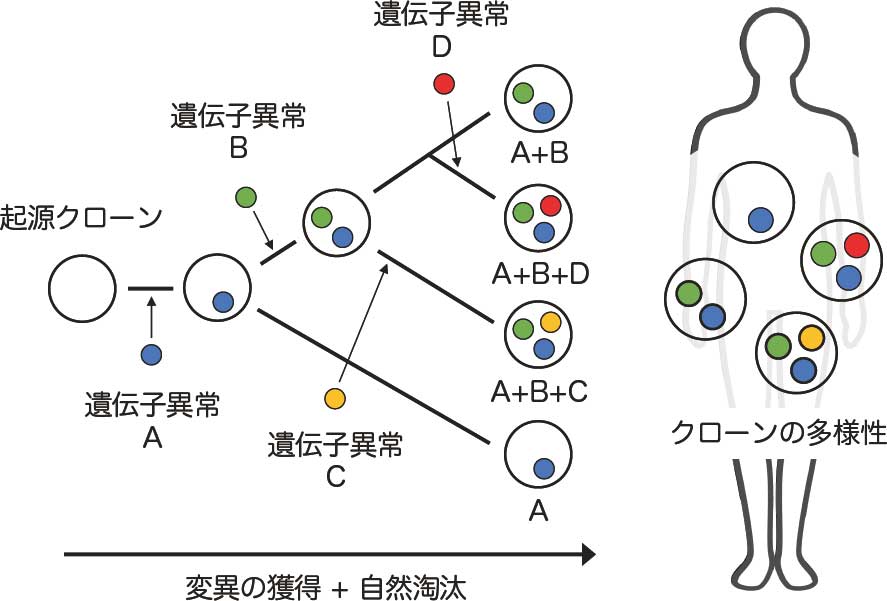
クローン進化(clonal evolution)とは、共通祖先クローンにゲノム異常やエピゲノム変化が確率的に蓄積し、分岐して生じたサブクローンが自然選択(natural selection)のもとで適応・拡大していく過程を指します。がんは、個体内で長い年月をかけてどのように多様性を獲得し、進化・適応するのでしょうか。この問いに答えることは、がんの発生・進展の理解と、診断・治療戦略の確立に直結します。
私たちは、前がん状態にある細胞集団やウイルス感染によって生じる多様な感染細胞集団が、いかにクローン進化して疾患に至るのかというメカニズムに関心を持ち、研究を進めています。多様性を高解像度で捉えるために、高深度シーケンスやシングルセル解析を駆使し、進化の軌跡と選択圧を明らかにします。
東京大学プレスリリース
主な参考論文
- Yamagishi et al., Nat Commun. 12, 4821, 2021
- Nagasaka et al., Proc. Natl. Acad. Sci. U S A., 17, 11685-11691, 2020
(4) データサイエンスを駆使した新たな治療薬開発研究
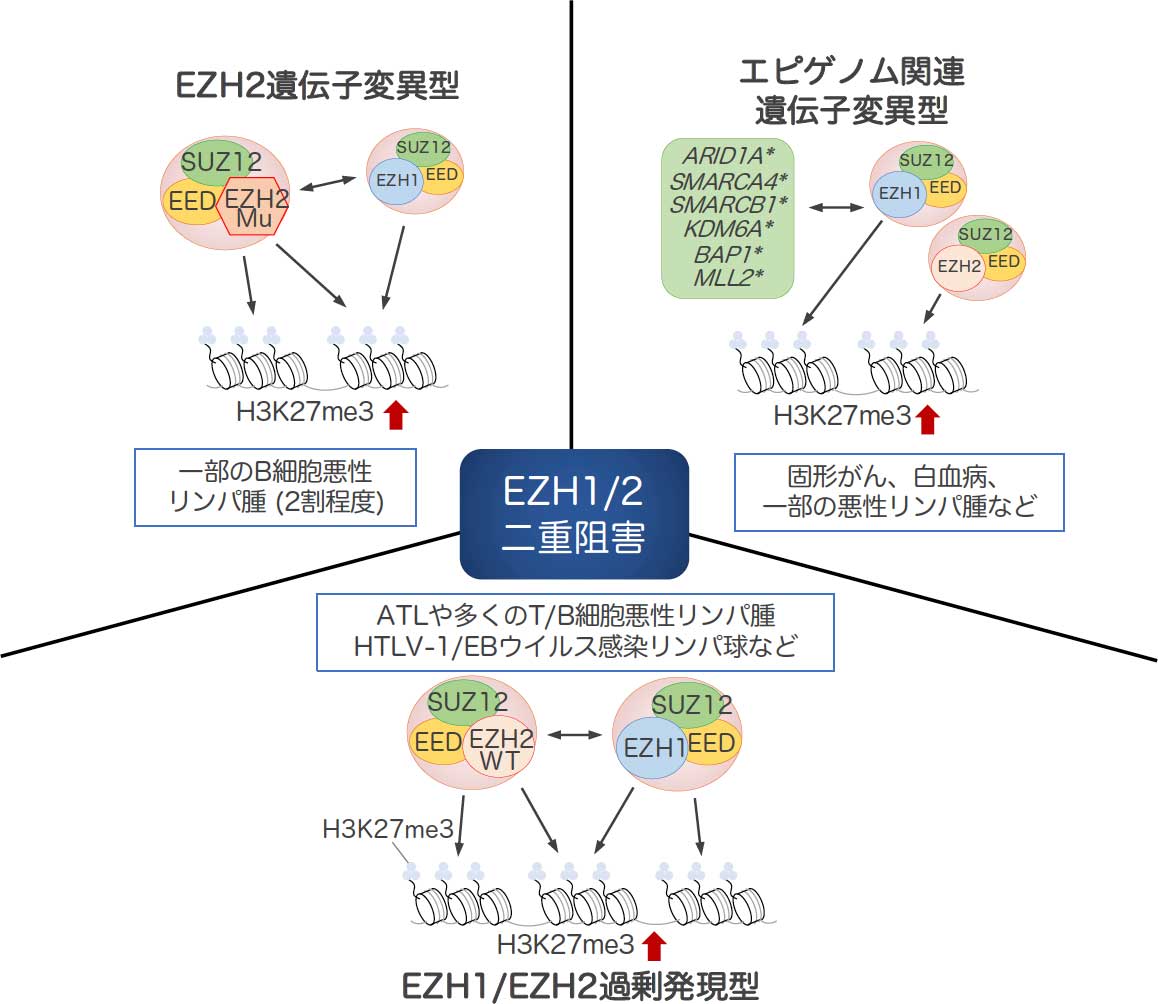
基礎研究の成果は、新たな診断・治療の創出につながります。私たちは、悪性リンパ腫やウイルス感染細胞のエピゲノム解析から、クロマチンの凝縮を駆動する酵素 EZH1/2(H3K27メチルトランスフェラーゼ)に着目し、製薬企業との産学連携により EZH1/2阻害薬 の開発に成功しました(Yamagishi et al., Nature, 2024)。この成果は、難治性疾患に対するエピゲノム標的治療の可能性を示すと同時に、臨床検体や疾患モデルを用いた分子病態・発症機序の解明という基盤研究の重要性を改めて私たちに教えてくれました。私たちは、基礎研究とデータサイエンスを通じて、新しい治療薬開発研究に力を入れ、研究しています。
東京大学プレスリリース
- https://www.k.u-tokyo.ac.jp/information/category/news/7778.html
- https://www.k.u-tokyo.ac.jp/information/category/press/10804.html
主な参考論文
- Yamagishi et al., 米国血液学会, 2022
- Yamagishi et al., Cell Rep., 29, 2321-2337, 2019
- Yamagishi M and Uchimaru K. Curr. Opin. Oncol. 29, 375-381, 2017
- Yamagishi et al., Nature, 627, 221-228,
(5) さまざまな感染症に関する研究 (東京大学UTOPIAとの共同研究)
東京大学 国際高等研究所 新世代感染症センター UTOPIAとの共同研究で、さまざまな感染症に対する基礎研究(免疫学、宿主とウイルスの相互作用、ウイルスゲノム進化、疫学など)を行っています。大学院で感染症研究を学びたい方はご連絡ください。
主な研究費 (研究代表者のみ)
国立研究開発法人日本医療研究開発機構 (AMED)
- 次世代がん医療加速化研究事業「治療反応性に関わる遺伝子翻訳及び代謝活性の腫瘍内不均一性の全体像解明と創薬シーズ探索」2024-2025年度
- 新興・再興感染症に対する革新的医薬品等開発推進研究事業「ゲノム情報を基盤としたHTLV-1感染症の病態形成機序の解明及び発症リスク予知アルゴリズム開発に関する総合的研究」2023-2025年度
- 新興・再興感染症研究基盤創生事業(多分野融合研究領域)「HTLV-1感染症のエピゲノムコードの解読と戦略的創薬を目指した基礎・臨床融合データサイエンス」2023-2025年度
- 新興・再興感染症研究基盤創生事業(多分野融合研究領域)「レトロウイルス病原性と持続感染の根源となる不均一な潜伏集団を生み出す宿主エピゲノム特性の解明と治療薬開発」2020-2022年度
- 革新的がん医療実用化研究事業「シングルセルゲノミクスを用いたNOTCH1変異クローンの特性解明及び新規治療法と層別化アルゴリズムの開発」2021-2023年度
- 創薬基盤推進研究事業「Indolent ATLの分子標的同定とEZH1/2阻害薬の適応拡大を目指した研究」2018-2020年度
科学研究費助成事業(科研費)
- 基盤研究(B)「がんエピゲノムの恒常性メカニズムの解明と持続的なエピゲノム治療法の確立」2024-2026年度
- 基盤研究(C)「ゲノム異常とエピゲノム異常によるATLの協奏的クローン進化メカニズムの解明」2021-2023年度
- 基盤研究(C)「腫瘍ウイルス感染初期に形成されるエピゲノム異常と発がん機構の解明に向けた統合解析」2018-2020年度
- 基盤研究(C)「がん等におけるシグナル異常活性化に関わるmiRNAとエピジェネティック因子の研究」2015-2017年度
- 若手研究(B)「ヒトレトロウイルスの潜伏化を制御する宿主細胞の多様性の分子機構」2012-2013年度